2016年7月6日から8月31日にDDBJデータ解析チャレンジを実施しました。DDBJデータ解析チャレンジでは、DDBJ保有のビッグデータである塩基配列公開データベースを用いて、チャレンジ課題の機械学習モデルの精度を競います。 構築モデルは京都大学のビッグデータ大学に投稿します。今回「DNA配列からのクロマチン特徴予測」のチャレンジ課題について、38名が参加して、延べ360回のモデル投稿がありました。モデル精度が上位1位~3位の最優秀賞・優秀賞・優良賞と、参加学生中で1位の学生賞の対象者を発表致します。また最優秀賞の構築モデルの概要を記載します。
DDBJデータ解析チャレンジ2016 入賞者
| 最優秀賞1st Prize of DDBJ Challenge Awards 2016 | 株式会社 情報数理バイオ 研究開発部 ライフサイエンスグループ望月 正弘 |
| 優秀賞2nd Prize of DDBJ Challenge Awards 2016 | 国立研究開発法人 理化学研究所 情報基盤センター バイオインフォマティクス研究開発ユニット松本 拡高(代表※)、尾崎 遼(※)※チームとして2名で参加 |
| 優良賞3rd Prize of DDBJ Challenge Awards 2016 | ビッツ株式会社岡山 利次 |
| 学生賞Student Prize of DDBJ Challenge Awards 2016 | 東京大学大学院 情報理工学系研究科 修士課程1年加藤 卓也 |
最優秀賞モデルの概要
望月氏のモデルは、Extremely Randomized Trees(ERT, 参考文献1) と Convolutional Neural Network(CNN, 参考文献2) の2種類の分類器を基盤として、Stacked Generalization(Stacking, 参考文献3) アンサンブル学習法で精度向上を図っています。特徴量は、チャレンジのクエリ配列だけでなく外部特徴量(ゲノム座標、遺伝子注釈情報)を組み入れています。1つ目のERTモデルはゲノム座標が特徴量でn(配列数) x m(染色体数)の行列です。ゲノム座標はクエリ配列をシロイヌナズナTAIR10ゲノム参照配列(参考文献 4)にアライメントして得ます。このERTモデルをGenomic Coordinates Based Model(GCBM)とします。2つ目のCNNモデルの特徴量は、クエリ配列と遺伝子注釈情報(TAIRからGFFファイルをダウンロード)です。Figure 1の様にforward/reverse strand別で遺伝子注釈情報を組み込んでいます。遺伝子注釈情報は定量値で、定義は次式になります。 変数rは減衰率で、変数dは遺伝子の1塩基目からの距離です。変数rが0なら特徴量は1になり、遺伝子中に対象塩基が含まれる事を表します。変数rが0より大きい時は、遺伝子開始塩基からの勾配値が与えられます。このCNNモデルをGene Annotated Sequences Based Model(GASBM)とします。Figure1: Structure of the neural network of GASBM
変数rは減衰率で、変数dは遺伝子の1塩基目からの距離です。変数rが0なら特徴量は1になり、遺伝子中に対象塩基が含まれる事を表します。変数rが0より大きい時は、遺伝子開始塩基からの勾配値が与えられます。このCNNモデルをGene Annotated Sequences Based Model(GASBM)とします。Figure1: Structure of the neural network of GASBM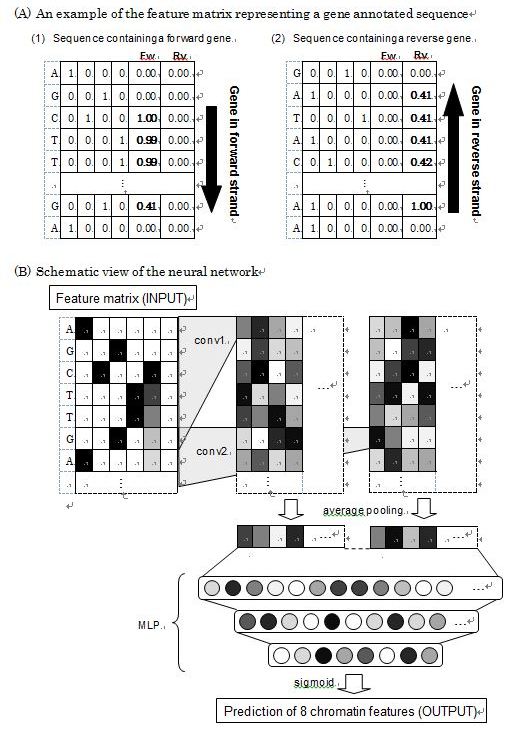 Figure2はベンチマーク結果です。ERT-GCBMモデルとCNN-GASBMモデルのパラメータ値などの詳細は、他の受賞者モデルも含めた全体解説(データクレンジング、特徴量選択、モデル訓練と予測法)と共に、報告論文で公開する予定です。Figure2: Benchmark result of models
Figure2はベンチマーク結果です。ERT-GCBMモデルとCNN-GASBMモデルのパラメータ値などの詳細は、他の受賞者モデルも含めた全体解説(データクレンジング、特徴量選択、モデル訓練と予測法)と共に、報告論文で公開する予定です。Figure2: Benchmark result of models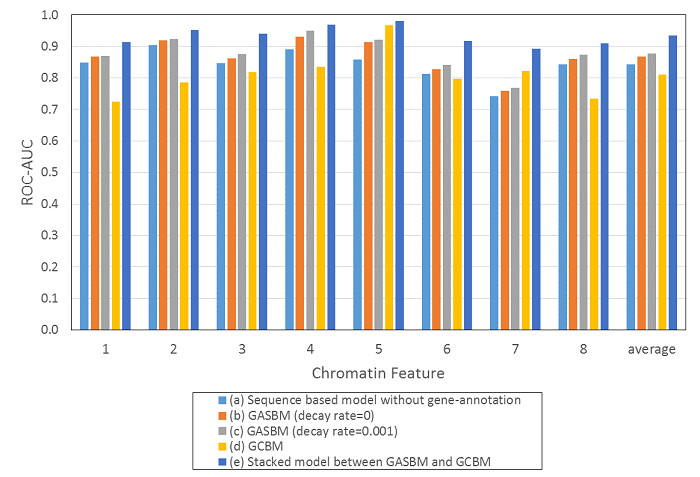 Reference[1] Geurts, P., Ernst, D. & Wehenkel, L. Extremely randomized trees. Machine Learning 63, 3-42 (2006).[2] LeCun, Yann, et al. “Gradient-based learning applied to document recognition.” Proceedings of the IEEE 86.11 (1998): 2278-2324.[3] Wolpert, D. H. Stacked generalization. Neural Networks 5, 241-259 (1992).[4] https://www.arabidopsis.org/
Reference[1] Geurts, P., Ernst, D. & Wehenkel, L. Extremely randomized trees. Machine Learning 63, 3-42 (2006).[2] LeCun, Yann, et al. “Gradient-based learning applied to document recognition.” Proceedings of the IEEE 86.11 (1998): 2278-2324.[3] Wolpert, D. H. Stacked generalization. Neural Networks 5, 241-259 (1992).[4] https://www.arabidopsis.org/
受賞モデルの精度結果
| DDBJ Challenge Award | AUC | Model Design | Tool Version |
|---|---|---|---|
| 1st Prize | 0.94564 | *2 Classifiers(Extremely Randomized Trees, CNN)*Ensemble Learning(Stacking)*External Data(Genomic Position, Gene Structure Annotation) | python=3.5scikit-learn=0.17.1chainer=1.10.0 |
| 2nd Prize | 0.89859 | *2 Classifiers(CNN, Product of Genomic Distance Decay Parameter and Nearest Training Data Output)*Ensemble Learning(Averaged)*External Data(Genomic Position) | julia=0.4.6python=2.7.10skflow(tensorflow=0.8.0) |
| 3rd Prize | 0.85428 | *7 Classifiers(Naive Bayes for Multivariate Bernoulli Models, Logistic Regression, Random Forest, Gradient Boosting, Extremely Randomized Trees, eXtreme Gradient Boosting, CNN)*Ensemble Learning (Stacking) | python=2.7.11numpy=1.10.4scikit-learn=0.17chainer=1.11.0xgboost=0.4a30 |
| Student Prize | 0.84318 | *3 Classifiers(LeNet like CNN, DeepBind like CNN, Variable filter DeepBind like CNN)*Ensemble Learning(Soft Voting) | python=2.7lasagne=0.2.dev1 |
お問い合わせ:DDBJへのお問い合わせの「DDBJデータ解析チャレンジ」からご連絡ください。