DDBJデータ解析チャレンジ
DDBJの大規模データを、遺伝研スパコンで解析しよう!!!
「DDBJデータ解析チャレンジ 2016」は終了いたしました。多くの方にご参加いただきありがとうございました。(受賞者の発表記事へ)
DDBJデータ解析チャレンジでは、DDBJ保有のビッグデータである塩基配列公開データベースを用いて、チャレンジ課題の機械学習モデルの精度を競います。 参加者がビッグデータ解析の経験を積める様に、学生やライフサイエンス専門外の方にも、遺伝研スーパーコンピュータの計算機環境を提供します。ビッグデータ解析ツールは、リクエストして頂ければスパコンへインストール致します。初日には参加者向けにキックオフ講習会を行い、課題やスパコンの使い方を説明します。皆様のご参加をお待ちしております。<h2>参加資格</h2>どなたでも参加可能です。ただし、遺伝研スーパーコンピュータ利用に関しては、アカウント発行基準に記載する利用資格により限定される場合がありますのでご注意ください。<h2>日程</h2>
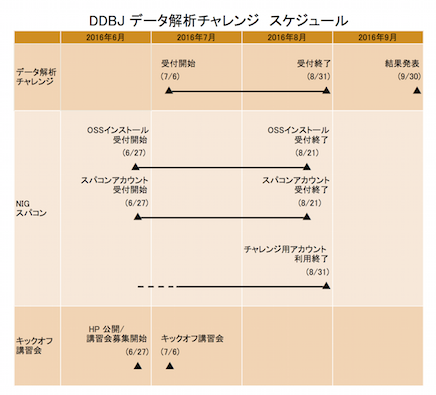
- 2016年6月27日(月):
- キックオフ講習会 参加受付開始、遺伝研スパコン期間利用申請、OSSインストール申請受付開始
- 2016年7月6日(水):
- 課題投稿の受付開始、キックオフ講習会
- 2016年8月21日(日):
- 遺伝研スパコン期間利用申請、OSSインストール申請受付の締切
- 2016年8月31日(水):
- 課題投稿の締切
- 2016年9月30日(金):
- 結果発表
チャレンジ課題
DNA配列からクロマチン特徴を予測します。DDBJ Sequence Read Archive(DDBJ SRA)からクロマチン特徴を注釈したデータベースに「ChIP-Atlas(九大沖博士、DBCLS大田博士)」があります。ChIP-Atlasデータベースに未掲載の生物種を対象として、DNA配列がクロマチン特徴領域か否かを予測します。
- 背景と意義
- ゲノム研究分野では、GWAS解析やQTL解析により疾患・病害リスクとなるDNA多型の同定が進められています。DNA多型の疾患・病害リスクの解釈には、オープンクロマチン領域情報、ヒストン修飾情報、転写因子結合部位情報などの遺伝子発現のオンオフに関わるクロマチン特徴情報が重要です。クロマチン特徴は組織条件が変わるとオンオフが変わります。一方、近年ゲノム機能解析データの蓄積や計算機性能の向上により、DNA配列からクロマチン特徴を組織条件を含めてモデル化を行い、全条件のクロマチン特徴の一挙予測が可能になりました。全条件モデルを組み合わせると、疾患・病害リスクと関連するDNA多型を数多くの候補の中から絞り込む事が可能になります。
- DDBJでは、高速DNAシークエンサ由来のビッグデータDDBJ SRAを提供しています。SRAを再解析したクロマチン特徴注釈データベース「ChIP-Atlas」を使えば、大規模なクロマチン特徴予測モデルの構築が可能になります。DNA多型候補の絞込み精度は、土台である遺伝子発現オンオフ特徴(=本チャレンジではクロマチン特徴)の精度に依存する為、高精度のクロマチン特徴予測モデルの構築が必要です。本チャレンジではChIP-Atlas注釈情報とDNA配列を用いて、高精度クロマチン特徴予測モデルの構築を目指します。
- データ説明と予測課題
- 課題データは、CellTypeクラスと Antigenクラスの組合わせ8条件で構成されています。8条件の訓練データを用いてモデルを構築し、テスト配列の予測結果を投稿して下さい。課題の8条件は、AntigenクラスはDNase-seq,Histone,RNA polymerase,TFs(Transcriptional factors)の4条件を含めて構成しています。生物種はChIP-Atlasデータベースに未掲載のモデル植物シロイヌナズナです。DNA配列ゲノム版は、TAIR10です。
- -----------------------------------入力訓練データ :60,000 DNA配列入力テストデータ:10,000 DNA配列出力訓練データ :8条件の正解(真偽)------------------------------------- <入力>1配列は、対象生物のゲノム上の200塩基(200個のACGTの並び)フラグメントで構成されています。1配列は、01コードで保存しており1行800データです。[例:AATGC...=10001000000100100100...]対応コード:A=1000,C=0100,G=0010,T=0001, その他の例外=0000<出力>出力訓練データ(正解データ)は01コードです。1が真で、DNA配列はクロマチン特徴領域を含む。0が偽でクロマチン特徴領域を含まない、に相当します。 <課題>入力テストデータの予測結果として、1万行8列で真の予測確率をビッグデータ大学に投稿して下さい。(列間区切はスペース)< /dd>
- チャレンジ効果
- 課題のクロマチン特徴予測モデルが高精度になれば、遺伝子発現オンオフに関わる(即ち疾患・病害リスクとなる)DNA多型の絞込み精度向上に繋がります。構築したシロイヌナズナのクロマチン特徴予測モデルは、他の植物へ応用可能な為に植物研究コミュニティに貢献する事が出来ます。
- データ置場
- (1) ビッグデータ大学内に設置(DDBJ-challenge.mat)
- (2) 遺伝研スーパーコンピュータ Phase2 の /home/challenge/data/DDBJ-challenge.mat
- データ形式
- .mat形式はMATLAB形式バイナリ ファイル (MAT ファイル)です。.mat形式の読込には、Rの場合:R.matlabのreadMat() , Pythonの場合:scipy.io.loadmat()をご利用ください。 </dl>
- 6月27日より利用申請を受け付けています。
- 遺伝研スパコン・アカウント申請
- 個別発行:6月27日から8月21日まで期間利用申請を こちら から受付します。利用目的の欄に「DDBJ Challenge」と記入ください。(記入例)
- 郵送でパスワードを送る為に、申請から利用開始まで2週間程かかります。
- チャレンジ参加者は2016年8月31日までの期間利用になります。ライフサイエンス研究にご利用の参加者は、チャレンジ期間後でも引き続き遺伝研スパコンをご利用頂けます。
- すでにNIGスパコンアカウントをお持ちの方:スパコンアカウントにDDBJ データ解析チャレンジのグループを作成しますのでこちらから申請してください。
- 遺伝研スパコン・OSSインストール申請
- 6月27日から8月21日までOSSインストール申請を こちら から受付します。
- 注意:インストールには申請から通常7~10日程かかります。計算機環境の条件によりインストールができない場合もございます。
- 遺伝研スパコンの基本的な利用方法につきましては、以下のサイトをご覧ください。
- ログイン接続、SGEジョブ投入方法
- プログラミング環境整備 (R, MATLAB, Python)
- Challenge参加者のみ利用可能です。
- MATLAB R2016a[含チャレンジ用toolbox]を、下記2通りで利用できます。
- (1) ローカルPCへのインストール
- (2) 遺伝研スパコンGPUノード
- ローカルPCへのMATLABダウンロードは こちらのサイト から申請して下さい。
- (学生に限らず申請可能)
- 申請の際は以下をご指定ください。
- University name:会社名・学校名と同じ内容を記入してください。
- Team name:個人のお名前、もしくはニックネームを入力してください。
- Team member:1 を入力してください。
- 場 所:国立研究開発法人 科学技術振興機構 東京本部別館 アクセス
- 主 催:日本DNAデータバンク(DDBJ)
- DDBJデータ解析チャレンジ
- DDBJお問い合わせ窓口
- 遺伝研スパコン利用、OSSインストールリクエストに関する質問
- 遺伝研スパコンお問い合わせ窓口
- DDBJデータ解析チャレンジの内容や結果は論文に投稿できますか?
- DDBJチャレンジ報告論文をジャーナルに投稿予定です。上位入賞者の方には、共著者としてご参加いただきます。詳しくは チャレンジ賞 を参照ください。
- 自分の PC に機械学習のためのプログラミング言語の開発環境を準備した方がいいですか?
- NIG スパコンには、R, MATLAB, Python などの機械学習ツールがインストールされていますので、NIG スパコンにログインして作業を行う場合は、ご自身のPC内に開発環境を作成する必要はありません。(NIG スパコンを利用する場合はアカウント申請が必要です。)ローカルPC で作業を行う場合には、ご自身で環境整備をして頂く必要があります。ローカルPCには期間中のみ無料利用できるMATLABをインストール可能です。
- チャレンジで利用した遺伝研スパコンのアカウントは、チャレンジ終了後どうなりますか?
- 利用終了後の9月1日から、アカウントは使用できなくなります。ライフサイエンス研究の為に使用したい方は、アカウント継続申請を こちら からリクエストしてください。継続申請フォームには下記を入力してください。 お問い合わせ種別:「その他」を選択してください。 お問い合わせ件名:「チャレンジアカウントの継続申請」を入力してください。
- 遺伝研スパコンの利用申請時に、フリーランス(または会社員)なので責任者を記入できないです。
- フリーランスの方は経歴情報を、会社員の方はご所属の会社情報を、遺伝研スパコンお問い合わせ窓口 までご連絡ください。
- Tutorialでは遺伝研スパコンのメモリ要求は24GBです。自分のプログラムではメモリエラーになりました。
- TutorialはMATLAB線形判別分析用のメモリ設定になっています。最大64GBまで設定可能 (-l mem_req=64G , -l s_vmem=64G) なので、プログラム毎に最適な値を指定してください。64GBを超えた値を設定すると、core dump 出力でジョブは強制終了しますので御注意ください。
- 配列データに0000コードを発見しました。また0000コードのみでクロマチン情報有の配列がありました。
- ゲノム領域の一部で塩基未決定の場合があり、課題では0000コードに設定しています。課題中の未決定塩基のみの配列は、あるピーク領域中の断片として、そのクロマチン情報が出力値に入っています。参加の皆様自身で、良い対処策を御考案下さい。
- challenge.q使用率が100%になっていて、使えなくて困っています。
- challenge.qノード数を16に変更しました。またリソースの占有を防ぐ為に、challenge.qの同時使用可能スロット数を150に変更しました。
- DDBJ-challenge.mat以外の外部データ利用は許可されていますか?
- 外部データ利用を許可していますが、下記の様に条件を設定します。 パラメータ(注1)の外部利用=可 転移学習用Pretrained Modelの利用=可(注2)(注1)=馬場博士スライドp.15参照
 (注2)=不正を防ぐ為に、外部入力配列は植物を禁止します
(注2)=不正を防ぐ為に、外部入力配列は植物を禁止します - Deep Learning用ライブラリを遺伝研スパコンへインストールするとエラーが出ます。
- UC Berkeleyの Caffe, Preferred Networks社 Chainer のインストール方法を スパコンHP に掲載しましたのでご参照ください。
- MATLABによる機械学習
- MATLAB によるディープラーニング (Web Seminar)
- Training a Deep Neural Network for Digit Classification – Example Code for MATLAB (英語)
- MATLABによる機械学習の基礎 ~特徴抽出・分類器・交差検定~ (Web Seminar)
- Mathworks Japan
- DDBJデータ解析チャレンジ期間中、MATLAB のライセンスを提供頂きます。 ライセンスの製品構成について