Japanese Genotype-phenotype Archive
JGA への登録
登録概要
JGA は DBCLS で承認された利用制限ポリシーを持つ、匿名化されたデータだけを受け付けています。登録者は DBCLS に JGA へのデータ提供を申請し、JGA は DBCLS から正式に提供が承認された連絡を受け、登録者に登録手順を案内します。
登録しようとしているデータの公開を制限すべきかどうかは、データを収集した個人との間で合意された協定から明らかなはずです。JGA に登録されたデータの公開は制限されます。JGA データを利用するためには、DBCLS に利用を申請し、承認される必要があります。
制限公開は論文公表までの間、データを非公開にすることとは異なります。 JGA を含む全ての DDBJ データベースでは、論文公表までの一定期間、データを非公開にすることができます。
メタデータオブジェクトに対して以下のプレフィックス付きアクセッション番号が割り振られます。
| プレフィックス | メタデータオブジェクト | 数字の桁数 | 例 |
|---|---|---|---|
| JGA | Submission | 6 | JGA000001 |
| JGAS | Study | 6 | JGAS000001 |
| JGAN | Sample | 9 | JGAN000000001 |
| JGAX | Experiment | 9 | JGAX000000001 |
| JGAR | Data | 9 | JGAR000000001 |
| JGAZ | Analysis | 9 | JGAZ000000001 |
| JGAD | Dataset | 6 | JGAD000001 |
| JGAP | Policy | 6 | JGAP000001 |
著者が論文中で引用すべき JGA アクセッション番号が指定されていない場合は、JGAS (study) 番号を引用することを推奨します。Study 番号は読者に研究の概要を示し、その Study に含まれているすべてのデータへのリンクを提供します。
アクセッション番号を引用する際の例文: 「Genotype data has been deposited at the Japanese Genotype-phenotype Archive (JGA, https://www.ddbj.nig.ac.jp/jga), which is hosted by the Bioinformation and DDBJ Center, under accession number JGASXXXXXX.」
塩基配列データ (raw/unaligned と aligned)
JGA でサポートしているファイル形式を以下に示します。 異なるファイル形式をもっている場合や不明な点がある場合は JGA に連絡してください。
データファイルが複数サンプルに由来するデータを含んでいる場合、投稿する前にファイルをサンプルごとに分割します。そうすることによって Run は1つのサンプルに由来するデータファイルだけを持つようになります。
BAM 形式
Binary Alignment/Map (BAM) ファイルは JGA への登録にとって好ましいファイル形式の一つです。BAM は Sequence Alignment/Map (SAM) ファイル形式のバイナリー圧縮です (詳細は SAMv1.pdf)。BAM ファイルは SAM/BAM 用ツール (例えば samtools) で human-readable なテキスト形式である SAM に変換することができます。BAM はアライメントされなかった unaligned read を含めることができます。 JGA はプライマリーデータとして Data に unaligned read を含む BAM を登録することを強く推奨しています。
圧縮という観点で BAM ファイルはほぼ最適化されているので、ファイルをさらに圧縮する必要はありません。
Fastq 形式
シングルとペアードの塩基配列データは下記の条件を満たした Fastq ファイルでの登録を推奨しています。
- Quality score が Phred スケールである。初期の Solexa パイプラインから出力された quality score は Phred スケールに変換されている必要があります。 ASCII とスペース区切りの数字で表された quality score をサポートしています。 Phred quality score のオフセットが 33 か 64 であるかは自動的に判定されます。
- Technical リード (アダプター、リンカー、バーコード配列) が取り除かれている。
- シングルリードは一つの Fastq ファイルで登録します。
- ペアードリードは分割し、二つの Fastq ファイルとして登録します。 リード名はペアの最初か二つ目のリードかを区別する ‘/1’ と ‘/2’ のような suffix を持っている必要があります。 リードの正規表現は “^(.*)([\\.|:|/|_])([12])$” になります。
- それぞれのリードが ‘@’ で始まる。
- ベースコールと quality score が ‘+’ で始める行で分けられている。
- Fastq ファイルは gzip か bzip2 で圧縮されている。
シングルリードを含む Fastq ファイルの例:
@read_name
GATTTGGGGTTCAAAGCAGTATCGATCAAATAGTAAATCCATTTGTTCAACTCACAGTTT
+
!''*((((***+))%%%++)(%%%%).1***-+*''))**55CCF>>>>>>CCCCCCC65
...
ペアードリードを含む Fastq ファイルの例:
@read_name/1
GATTTGGGGTTCAAAGCAGTATCGATCAAATAGTAAATCCATTTGTTCAACTCACAGTTT
+
!''*((((***+))%%%++)(%%%%).1***-+*''))**55CCF>>>>>>CCCCCCC65
@read_name/2
GATTTGGGGTTCAAAGCAGTATCGATCAAATAGTAAATCCATTTGTTCAACTCACAGTTT
+
!''*((((***+))%%%++)(%%%%).1***-+*''))**55CCF>>>>>>CCCCCCC65
...
SFF 形式
454 プラットフォームからのデータは、SFF での登録が推奨されます。
圧縮という観点で SFF ファイルはほぼ最適化されているので、ファイルをさらに圧縮する必要はありません。
アレイデータ (遺伝子型タイピング、SNP、遺伝子発現)
JGA は全ての種類のアレイプラットフォームからのデータを受け付けています。例えば、genotypes、遺伝子発現やメチル化解析データなどです。
JGA はこれらのデータの登録に柔軟に対応しており、Analysis にアレイ実験から得られたあらゆる種類のデータファイルを格納することができます。
JGA は生データ (例 CEL) と解析したデータの両方を登録することを推奨します。これらは利用者が結果をみるためだけではなく、論文で示されている結果を再現するためにも役立ちます。
JGA はサンプルに関連した表現型 (phenotype) 情報も Analysis にアーカイブしています。
変異データ
JGA は variation データの VCF 形式での登録を推奨しています。JGA Analysis に vcf ファイルを登録することができます。
その他のデータ
Analysis は複数の Data もしくは Sample を参照することができるので、これらのオブジェクトに登録されているデータを解析、集計したデータを Analysis に登録することができます。Analysis に登録できるファイルの例を以下にリストします。
- 複数 Data に登録された配列データから作成したリファレンス配列 GFF3 ファイル
- 複数 Analysis に登録されたアレイデータを正規化し表にまとめたファイル
メタデータ
JGA データモデルは Sequence Read Archive のモデルを拡張してつくられています。JGA メタデータは XML オブジェクトで構成されています。
JGA XML schema: JGA xsd
公開される項目
JGA メタデータのうち以下のオブジェクトは登録の概要を示すため、データの提供開始後に公開されます。
- Study:
- 全ての内容
- Dataset:
- 全ての内容と含まれる Sample 数とファイルサイズ。
- Policy:
- 全ての内容
Submission
登録プロセスを管理します。登録者の連絡先情報を含みます。
XML schema: JGA.submission.xsd
Study
研究を記述します。 研究のタイトル、study type と論文に使われるようなアブストラクトを含みます。論文公表後、登録したデータを使った論文の PubMed ID を追加し、論文情報を更新します。
XML schema: JGA.study.xsd
Sample
研究で使った各サンプルを記載します。一般的に Sample は個人に対応します。 必須項目は少ないですが、できるだけ詳細な情報を記入し、研究対象者の表現型を含むサンプルについての有用な情報を提供することを推奨します。
XML schema: JGA.sample.xsd
Experiment
実験のセットアップを記述します。サンプルの調整からデータの取得にいたるまでのプロトコールや使ったシークエンサを記載します。次世代シークエンス実験にのみ対応しています。
XML schema: JGA.experiment.xsd
Data
次世代シークエンスデータについての情報と、それらと Experiment との関係を記述します。ファイルをまとめる役割を果たします。
XML schema: JGA.data.xsd
Analysis
アレイデータ、データを解析、集計した結果を登録します。 変異データは VCF ファイルとして登録することができます。 それぞれの Analysis には一つの VCF ファイルしか登録できません。また、VCF で使われたサンプルは JGA Sample に対応している必要があります。 使用したリファレンス配列を INSDC のアクセッション番号、もしくは広く使われているラベル名で記載します。
XML schema: JGA.analysis.xsd
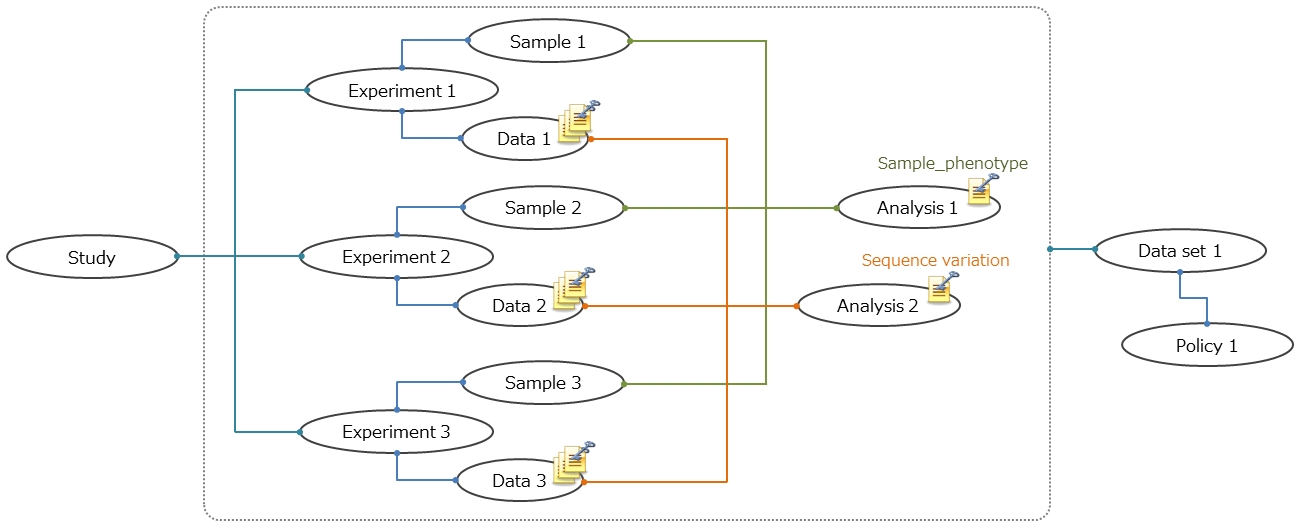
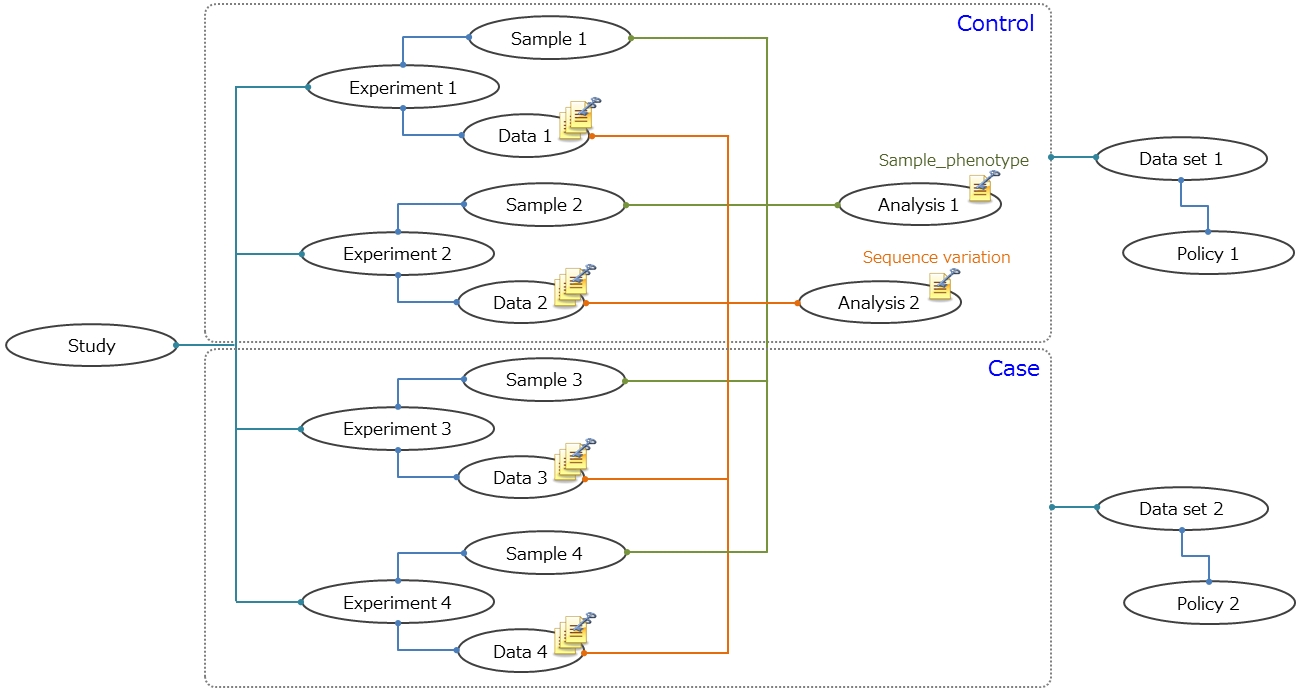
Dataset
Data XML と Analysis XML に記載されているデータファイルを,データセットという単位にまとめます。 Study 中に適用されるポリシー (利用制限事項) が異なるデータ (Control と Case 等) が含まれている場合、データセットを分ける必要があります。
一般的にポリシーは個人 (Sample) との間のインフォームドコンセントによって規定されます。
XML schema: JGA.dataset.xsd
Policy
データ利用制限ポリシーを記載します。NBDC ポリシー以外に適用すべきポリシーがなければ作成する必要はありません。
NBDC ポリシー以外に適用すべきポリシーがある場合は、DBCLS にポリシーを登録し、発行される JGAP 番号を Dataset から参照します。
XML schema: JGA.policy.xsd
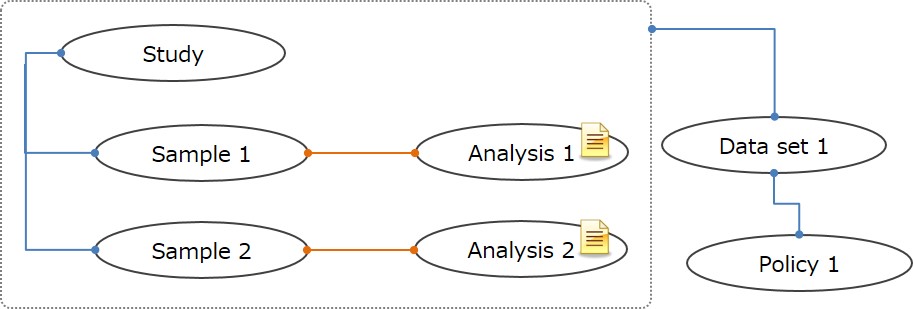
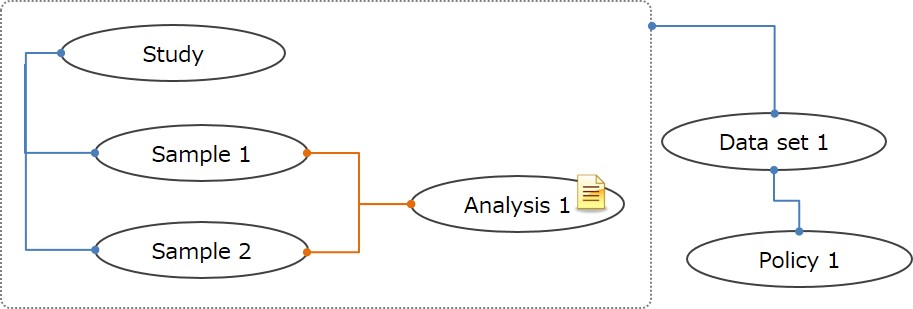
Analysis のみの登録
集計されたデータ、もしくは、Experiment-Data に格納するのが適切ではないデータを登録する場合は Sample-Experiment-Data ではなく Sample-Analysis セットでデータを登録することができます。
Sample-Analysis セットでデータ登録をする場合は JGA チームに連絡します。
登録の更新
登録を更新するためには JGA チームに連絡します。
公開済み Study/Dataset にデータを追加する場合、追加データを非公開にすることはできません。公開してもよくなった時点で追加・公開する、もしくは、追加分は Study を分けて非公開にしてください。
データの公開
NBDC ヒトデータベースで JGA データに対応する hum 番号の専用サイトが公開されると、JGA データも公開されます。