BLAST
DDBJ では,Web版 BLAST の他に,以下の方法でも BLAST サービスを提供しています。
Program:プログラム
解析の用途に合わせ,以下のプログラムのいずれかを左カラムより選択します。
デフォルトでは,blastn が設定されてます。megablast は新たに追加されたプログラムです。
| プログラム | クエリ | データベース | 説明 | |
|---|---|---|---|---|
| megablast | 塩基配列 | 塩基配列 | あなたの塩基配列を塩基配列データベースと比較します。 長大な塩基配列で相同性検索を行いたい場合,blastn より高速に検索結果が得られます。 |
|
| blastn | 塩基配列 | 塩基配列 | あなたの塩基配列を塩基配列データベースと比較します。 | |
| tblastn | アミノ酸配列 | 塩基配列 | あなたのアミノ酸配列に対して,塩基配列データベースを表裏合わせて6通りの読み枠で翻訳しながら比較します。 | |
| tblastx | 塩基配列 | 塩基配列 | あなたの塩基配列を表裏合わせて6通りの読み枠で翻訳しながら,同様に翻訳された塩基配列データベースと比較します。 | |
| blastp | アミノ酸配列 | アミノ酸配列 | あなたのアミノ酸配列をアミノ酸配列データベースと比較します。 | |
| blastx | 塩基配列 | アミノ酸配列 | あなたの塩基配列を表裏合わせて6通りの読み枠で翻訳しながら,アミノ酸配列データベースと比較します。 |
Query:検索配列名,検索配列データ
- FASTA 形式で検索配列を入力して下さい。
- データを直接テキストボックスに入力するか,データを格納したファイルを指定(ファイルアップロード機能)します。
- 検索する配列が複数の場合は,各配列を区別するために配列名が必要です。先頭に「>」をつけた配列名の行を各配列の前においてください (multi FASTA 形式)。
- 検索する配列がひとつの場合は,配列名は必要ありません。配列名をつける場合は,先頭に「>」をつけた配列名の行を配列の前において下さい。
FASTA 形式
>my query sequence 1
CACCCTCTCTTCACTGGAAAGGACACCATGAGCACGGAAAGCATGATCCAGGACGTGGAA
GCTGGCCGAGGAGGCGCTCCCCAGGAAGACAGCAGGGCCCCAGGGCTCCAGGCGGTGCTG
GTTCCTCAGCCTCTTCTCCTTCCTGCTCGTGGCAGGCGCCGCCAC
複数配列の入力例 (multi FASTA 形式)
>>my query sequence 1
CACCCTCTCTTCACTGGAAAGGACACCATGAGCACGGAAAGCATGATCCAGGACGTGGAA
GCTGGCCGAGGAGGCGCTCCCCAGGAAGACAGCAGGGCCCCAGGGCTCCAGGCGGTGCTG
GTTCCTCAGCCTCTTCTCCTTCCTGCTCGTGGCAGGCGCCGCCAC
>my query sequence 2
GGCCAGGGCACCCAGTCTGAGAACAGCTGCACCCGCTTCCCAGGCAACCTGCCTCACATG
CTTCGAGACCTCCGAGATGCCTTCAGCAGAGTGAAGACTTTCTTTCAAATGAAGGATCAG
CTGGACAACATATTGTTAAAGGAGTCCTTGCTGGAGGACTTTAAG
>my query sequence 3
ATGGGTCTCACCTCCCAACTGCTTCCCCCTCTGTTCTTCCTGCTAGCATGTGCCGGCAAC
TTTGCCCACGGACACAACTGCCATATCGCCTTACGGGAGATCATCGAAACTCTGAACAGC
CTCACAGAGCAGAAGACTCTGTGCACCAAGTTGACCATAACGGAC
配列サイズが巨大な場合,配列数が多い場合などは,以下のような理由で結果を正常に取得出来ない場合があります。
そのような場合は,配列数を少なくするか配列を短くする等して再度お試し下さい。
- 結果が巨大になりブラウザで表示出来ない
- 検索時間が長くなり過ぎてタイムアウトする
Data Sets {#data sets}
塩基配列(データベース,DIVISION)
データベース
検索対象となるデータベースを指定します。 以下の選択肢からひとつを選びます。
| 塩基配列データベース | |
|---|---|
| DDBJ ALL | DDBJ 定期リリース + 新着データ |
| DDBJ New | DDBJ 定期リリース後の新着データ |
| 16S rRNA | DDBJ 定期リリースから 16S rRNA 配列を取り出したデータ |
| RefSeq NA | RefSeq (Genomics + RNA) |
DIVISION (DDBJ ALL / DDBJ New)
DDBJ ALL または DDBJ New を選択した場合,以下の選択肢から,検索対象とする DIVISION の指定が可能です(複数可)。デフォルトでは、Standard divisions のうち SYN, ENV を除いた 10 divisions が選択されています。 EST については登録数の多い21の生物種から選択することが出来ます。
| Standard divisions | ||
|---|---|---|
| Human | HUM | ヒト |
| Primates | PRI | 霊長類 (ヒトを除く) |
| Rodents | ROD | 齧歯類 |
| Mammals | MAM | 哺乳類 (ヒト、霊長類、齧歯類を除く) |
| Vertebrates | VRT | 脊椎動物 (ヒト、霊長類、齧歯類、哺乳類を除く) |
| Invertebrates | INV | 無脊椎動物 |
| Plants | PLN | 植物・酵母・藻類 |
| Bacteria | BCT | バクテリア(原核生物) |
| Viruses | VRL | ウイルス |
| Phages | PHG | バクテリオファージ |
| Synthetic DNAs | SYN | 合成配列 (合成遺伝子) |
| ENV | ENV | 環境サンプル (environmental samples) |
| High throughput divisions | ||
|---|---|---|
| HTC | HTC | High Throughput cDNAs |
| HTG | HTG | High Throughput Genomic sequences |
| TSA | TSA | Transcriptome Shotgun Assembly |
| EST divisions | |
|---|---|
| A.thaliana | Arabidopsis thaliana (シロイヌナズナ) |
| B.taurus | Bos taurus (ウシ) |
| C.elegans | Caenorhabditis elegans (線虫) |
| C.reinhardtii | Chlamydomonas reinhardtii (クラミドモナス) |
| C.intestinalis | Ciona intestinalis (カタユウレイボヤ) |
| D.rerio | Danio rerio (ゼブラフィッシュ) |
| D.discoideum | Dictyostelium discoideum (キイロタマホコリカビ) |
| D.melanogaster | D.melanogaster (ショウジョウバエ) |
| G.gallus | Gallus gallus (ニワトリ) |
| G.max | Glycine max (ダイズ) |
| H.sapiens | Homo sapiens (ヒト) |
| H.vulgare | Hordeum vulgare (亜種も統合) |
| M.truncatula | Medicago truncatula (特殊ライブラリも統合) |
| M.musculus | Mus musculus (ハツカネズミ) |
| O.sativa | Oryza sativa (亜種レベルも統合) |
| R.norvegicus | Rattus norvegicus (Rattus sp. も統合) |
| S.lycopersicum | Solanum lycopersicum (トマト) |
| T.aestivum | Triticum aestivum (コムギ) |
| X.laevis | Xenopus laevis (アフリカツメガエル) |
| X.tropicalis | Xenopus tropicalis (アフリカツメガエル) |
| Z.mays | Zea mays (トウモロコシ) |
| Others | 上記以外 (Others) |
| その他 | ||
|---|---|---|
| Patent | PAT | 特許データ |
| Unannotated Seq | UNA | 未注釈データ |
| GSS | GSS | ゲノム研究関連 (genome survey sequences) |
| STS | STS | sequence tagged sites |
*特許データの利用に際しては,特許データ利用に関する注意をご参照ください。
データベースオプション(RefSeq)
| 定期リリース(genomic/RNA) | |
|---|---|
| Fungi | 菌類 |
| Invertebrate | 無脊椎動物 |
| Microbial | 細菌 |
| Mitochondrion | ミトコンドリア |
| Plant | 植物 |
| Plasmid | プラスミド |
| Plastid | 色素体 |
| Protozoa | 原生生物 |
| Vertebrate Mammalian | 哺乳類 |
| Vertebrate Other | 哺乳類以外の脊椎動物 |
| Viral | ウィルス |
| Daily Updates | Daily Updates |
| モデル生物(Genomic) | |
|---|---|
| H. sapiens | ヒト |
| モデル生物(RNA) | |
|---|---|
| B. taurus | ウシ |
| D. rerio | メダカ |
| H. sapiens | ヒト |
| M. musculus | マウス |
| R. norvegicus | ラット |
| X. tropicalis | アフリカツメガエル |
アミノ酸配列(データベース,DIVISION)
データベース
デフォルトは UniProt(Swiss-Prot + TrEMBL)です。
| Proterin Databases | |
|---|---|
| UniProt (Swiss-Prot + TrEMBL) | Swiss-Prot + TrEMBL |
| UniProt (Swiss-Prot) | Swiss-Prot |
| UniProt (TrEMBL) | TrEMBL |
| Patent | JPO,EPO,USPTO,KIPO の全データ (Patent を選択すると4つ全てにチェックが入ります。個々の選択は可能です。不要なチェックを外してください。) |
| DAD (periodical release + daily updates) | DAD 定期リリース + 新着データ |
| DAD (daily updates) | DAD 定期リリース後の新着データ |
| RefSeq AA | Refseq(Protein) |
*特許データの利用に際しては,特許データ利用に関する注意をご参照ください。
*現在のバージョンは こちら からご確認ください。
DIVISION(DAD)
検索対象とする DIVISION を指定します。以下の選択肢から指定が可能です(複数可)。デフォルトでは、Standard divisions のうち SYN, ENV を除いた 10 divisions が選択されています。 EST については登録数の多い21種のデータから選択することが出来ます。
| Standard divisions | ||
|---|---|---|
| Human | HUM | ヒト |
| Primates | PRI | 霊長類 (ヒトを除く) |
| Rodents | ROD | 齧歯類 |
| Mammals | MAM | 哺乳類 (ヒト、霊長類、齧歯類を除く) |
| Vertebrates | VRT | 脊椎動物 (ヒト、霊長類、齧歯類、哺乳類を除く) |
| Invertebrates | INV | 無脊椎動物 |
| Plants | PLN | 植物・酵母・藻類 |
| Bacteria | BCT | バクテリア(原核生物) |
| Viruses | VRL | ウイルス |
| Phages | PHG | バクテリオファージ |
| Synthetic DNAs | SYN | 合成配列 (合成遺伝子) |
| ENV | ENV | 環境サンプル (environmental samples) |
| High throughput divisions | ||
|---|---|---|
| HTC | HTC | High Throughput cDNAs |
| HTG | HTG | High Throughput Genomic sequences |
| TSA | TSA | Transcriptome Shotgun Assembly |
| EST divisions | |
|---|---|
| A.thaliana | Arabidopsis thaliana (シロイヌナズナ) |
| B.taurus | Bos taurus (ウシ) |
| C.elegans | Caenorhabditis elegans (線虫) |
| C.reinhardtii | Chlamydomonas reinhardtii (クラミドモナス) |
| C.intestinalis | Ciona intestinalis (カタユウレイボヤ) |
| D.rerio | Danio rerio (ゼブラフィッシュ) |
| D.discoideum | Dictyostelium discoideum (キイロタマホコリカビ) |
| D.melanogaster | D.melanogaster (ショウジョウバエ) |
| G.gallus | Gallus gallus (ニワトリ) |
| G.max | Glycine max (ダイズ) |
| H.sapiens | Homo sapiens (ヒト) |
| H.vulgare | Hordeum vulgare (亜種も統合) |
| M.truncatula | Medicago truncatula (特殊ライブラリも統合) |
| M.musculus | Mus musculus (ハツカネズミ) |
| O.sativa | Oryza sativa (亜種レベルも統合) |
| R.norvegicus | Rattus norvegicus (Rattus sp. も統合) |
| S.lycopersicum | Solanum lycopersicum (トマト) |
| T.aestivum | Triticum aestivum (コムギ) |
| X.laevis | Xenopus laevis (アフリカツメガエル) |
| X.tropicalis | Xenopus tropicalis (アフリカツメガエル) |
| Z.mays | Zea mays (トウモロコシ) |
| Others | 上記以外 (Others) |
| その他 | ||
|---|---|---|
| Patent | PAT | 特許データ |
| Unannotated Seq | UNA | 未注釈データ |
| GSS | GSS | ゲノム研究関連 (genome survey sequences) |
| STS | STS | sequence tagged sites |
データベースオプション(RefSeq)
| 定期リリース(Protein) | |
|---|---|
| Fungi | 菌類 |
| Invertebrate | 無脊椎動物 |
| Microbial | 細菌 |
| Mitochondrion | ミトコンドリア |
| Plant | 植物 |
| Plasmid | プラスミド |
| Plastid | 色素体 |
| Protozoa | 原生生物 |
| Vertebrate Mammalian | 哺乳類 |
| Vertebrate Other | 哺乳類以外の脊椎動物 |
| Viral | ウィルス |
| Daily Updates | Daily Updates |
| モデル生物(RNA) | |
|---|---|
| B. taurus | ウシ |
| D. rerio | メダカ |
| H. sapiens | ヒト |
| M. musculus | マウス |
| R. norvegicus | ラット |
| X. tropicalis | アフリカツメガエル |
Optional Parameters
SCORES:検索結果一覧の表示数
相同性スコアの順位表を,第何位まで表示するかを指定します。
デフォルトは100位までです。
検索結果に期待される対象が含まれない場合,この値をより大きな値にすると,結果が改善される可能性があります。
ALIGNMENTS:整列 (アライメント)
相同な配列との整列を,相同性スコアの順で第何位まで表示させるかを指定します。
検索結果に期待される対象が含まれない場合,この値をより大きな値にすると,結果が改善される可能性があります。
EXPECT:期待値(E-value) {#expect value}
出力される配列に関して統計的な閾値を指定します。デフォルトは10ですが,これは偶然に一致する配列が10本あるという条件を意味しており,この条件よりも偶然に一致する配列が少ないと統計的に計算された配列が出力されます。
多くの相同な配列を出力させるには,この値を大きく指定します。逆に,非常に高い相同性を持つ配列だけを出力させるには,この値を小さく指定します。
また,指数表記で指定可能です。(例:1.0E+1)
SCORING MATRIX:アミノ酸置換表 {#scoring matrix}
アミノ酸配列の置換行列の表を指定します。
blastx, blastp, tblastn, tblastx で指定可能です。指定可能な置換行列は以下のとおりです。
デフォルトでは BLOSUM62 が設定されます。
| PAM30 | PAM30 substitution matrix |
| PAM70 | PAM70 substitution matrix |
| PAM250 | PAM250 substitution matrix |
| BLOSUM45 | BLOSUM Clustered Scoring Matrix |
| BLOSUM50 | BLOSUM Clustered Scoring Matrix |
| BLOSUM62 | BLOSUM Clustered Scoring Matrix |
| BLOSUM80 | BLOSUM Clustered Scoring Matrix |
| BLOSUM90 | BLOSUM Clustered Scoring Matrix |
FILTER:フィルター
検索配列データのフィルタリング(マスキング)を指定します。デフォルトではフィルタリングを行います。
フィルタリングにより,問い合わせ配列のうち構造の複雑度が低い領域を無視することができます。
一般に proline-rich 領域や poly-A tail などの領域は構造的な偏りが反映されて異常に高いスコアで一致する傾向がありますが,フィルタリングによりそのような有意でない一致を結果から除くことができます。
blastn 使用時には Tatusov and Lipman によるDUSTプログラムにより,他の場合は Wootton & Federhen (Computers and Chemistry, 1993) による SEG プログラムによりフィルタリングされます。
フィルタプログラムによって見つけられた低複雑度領域は,塩基配列の場合 “N”,アミノ酸配列の場合 “X” の文字で置き換えられます。
WORD SIZE {#word size}
自然数を入力します。デフォルトでは blastn の megablast の場合は 28,blastn の blastn の場合は11,
blastp,blastx,tblastn,tblastx の場合は 3 です。
blastn の場合は,wordsize を 11 以外の数に変えるべきではありません。
Request ID,検索結果表示
Request ID {#request id}
検索実行後の画面に Request ID が表示されますので,必ず控えて下さい。(問い合わせや Result Viewer (後述)で結果を見たい時などに必要です)
Request ID:wabi_blast_2013-0314-1407-23-16-946732
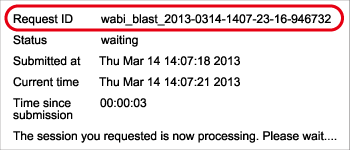
結果画面に表示される情報 {#result screen}
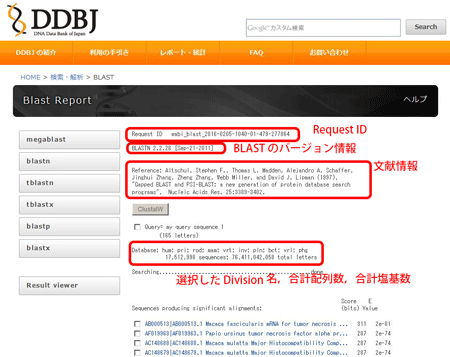
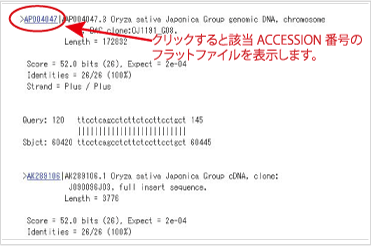
結果中のアクセッション番号の検索・表示 {#view flatfile}
該当するアクセッション番号にチェックを入れて、”getentry” ボタンをクリックすると、getentry の結果が表示されます。
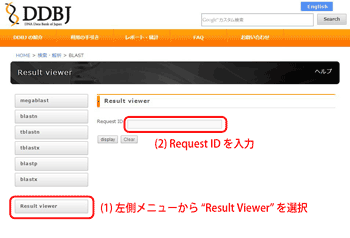
検索結果の閲覧 {#result viewer}
Result Viewer
ブラウザを閉じてしまっても、Result Viewer を用いてRequest ID から結果を閲覧することができます。
検索結果閲覧期間は、実行後7日間です。
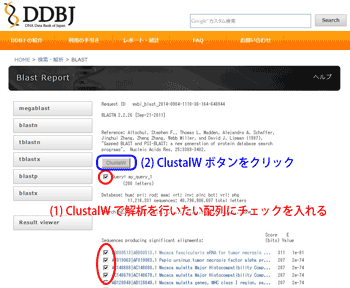
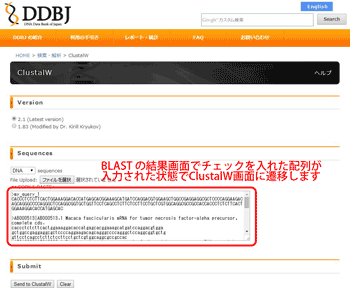
ClustalW Set up
チェックボックスで ClustalW で解析を行いたい配列を選択して”ClustalW” ボタンをクリックすると、選択した配列が入力された状態でClustalW 画面が開きます。
1. 2.
2.
原著論文,関連論文および参考文献
原著論文
- Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ. (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 25(17):3389-3402.
関連論文
- Zhang J, Madden TL. (1997) PowerBLAST: A New Network BLAST Application for Interactive or Automated Sequence Analysis and Annotation. Genome Res.7(6):649-656.
- Madden TL, Tatusov RL, Zhang J. (1996) Applications of network BLAST server. Methods Enzymol. 266:131-141.
- Gish W, States DJ. (1993) Identification of protein coding regions by database similarity search. Nat Genet. 3(3):266-272.
- Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. (1990) Basic local alignment search tool. J Mol Biol. 215(3):403-10.
- Karlin S, Altschul SF. (1990) Methods for assessing the statistical significance of molecular sequence features by using general scoring schemes. Proc Natl Acad Sci U S A. 87(6):2264-2268.
参考文献
- 「改訂 第2版 バイオデータベースとウェブツールの手とり足とり活用法」(羊土社)
編集/中村保一,石川 淳,磯合 敦,平川美夏,坊農秀雅 ISBN978-4-7581-0811-9 - [BLAST] Ian Korf, Mark Yandell and Joseph Bedell, OREILLY