Genomic Expression Archive
アレイデザイン
Array design format (ADF) ファイル
ADF (Array Design Format) ファイルはマイクロアレイチップに関する情報を記述するファイルです。ADF は [main] タグで区切られた二つのセクションから構成されます。
- ヘッダーセクション: 登録者、プローブやプローブセットのデザイン方法等のアレイ全体に関するメタデータ
- テーブルセクション: 各行にプローブ/プローブセット、各列にアノテーションを記載。例としてはユニークなプローブ ID、プローブ配列、プローブのスポットアレイにおける位置、プローブのリファレンスゲノムにおける位置、他のデータベースエントリへの参照など。
ADF ファイルはタブ区切りテキストファイル形式 (*.txt) でスプレッドシートプログラムで閲覧、編集することができます。
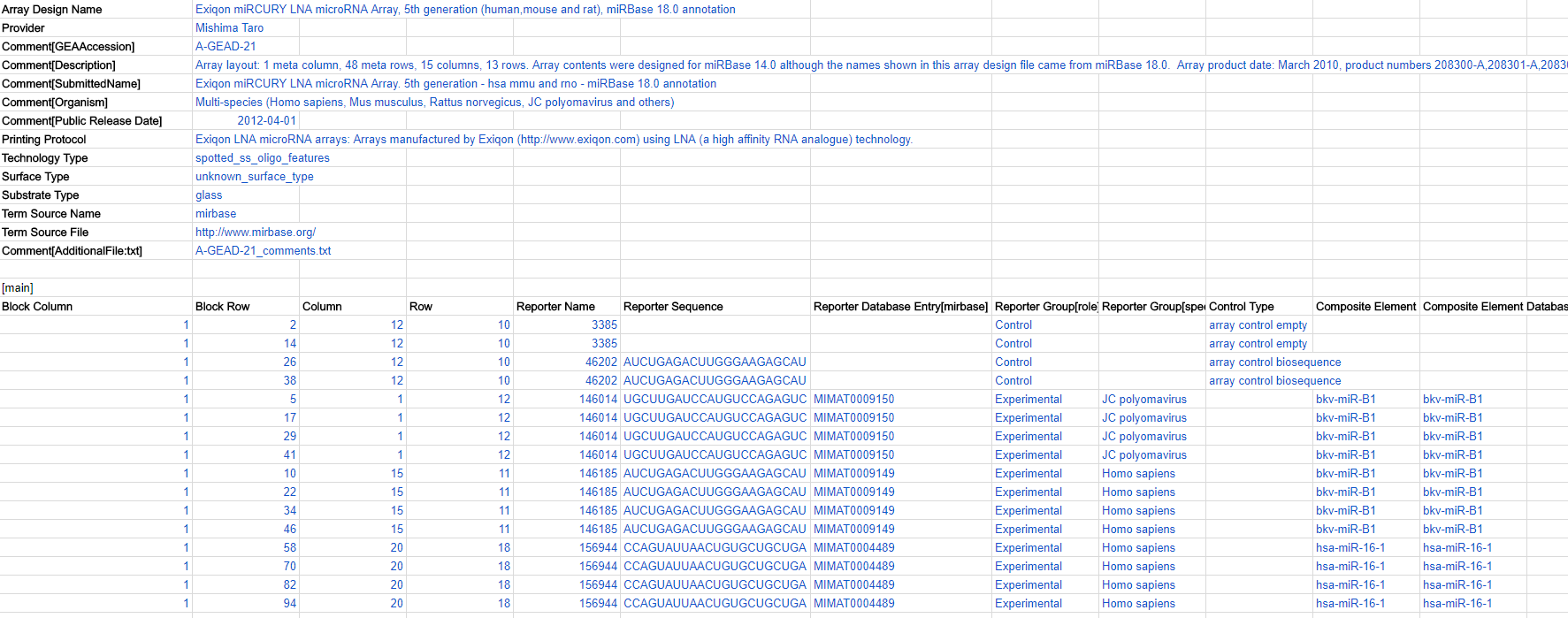
ADF の例(一部):
ADF メタデータヘッダー
各行(フィールド)はヘッダー(下の例では太字)で始めます。ヘッダーは controlled vocabulary を一回のみ記載します。GEA が提供している ADF テンプレートにはヘッダーが埋め込まれているのでゼロから作成する必要はありません。テンプレート中のヘッダーは編集せず各フィールドに可能な限り内容を記載してください(下の例では青字)。
ADF ヘッダーフィールドリスト (* 必須フィールド):
| No. | ADF header field | What is it? | Allowed values | Example |
|---|---|---|---|---|
| 1 | * Array Design Name | アレイデザインの内容が示すタイトル。製造業者 (例 Agilent) やラボの名称、対象生物種、バージョン番号、アレイの用途 (例 genotyping)、プローブ/フィーチャーの数 (例 450k) を含めてください。 | Agilent human micoRNA microarray miRBase Release 14.0, 8x15k (GridName 029297, 82 cols x 192 rows) | |
| 2 | Version | アレイデザインのバージョン番号。 | version 3.0 | |
| 3 | * Provider | アレイデザインの登録者の名前。 | Mishima Taro | |
| 4 | * Comment[Organism] | プローブ配列の由来生物種。 | NCBI Taxonomy に登録されている生物名(学名)を記載。複数生物種を対象としたアレイの場合は生物名をセミコロン;で区切って列挙。 | Homo sapiens |
| 5 | * Comment[Description] | マイクロアレイのデザイン方法と用途に関する簡潔な説明。可能であれば安定したオンラインリソースの URL を含めます。 | This custom commercial microarray consists of 84881 (86205 total including 1324 control probes) 60-mer oligonucleotide probes derived from the genomes and EST database of Emiliania huxleyi strain (… etc…) | |
| 6 | * Comment[Public Release Date] | アレイデザインを非公開に保つ場合は “Hold” と記入。即日公開の場合は登録日を記入。 | 登録日は YYYY-MM-DD 形式で記載 | 2014-04-25 |
| 7 | Printing Protocol | プローブのアレイへのプリント方法を記載。可能であれば安定したオンラインリソースの URL を含めます。 カスタムアレイを商業的な方法以外で作成した場合に重要な説明になります。 | The 60-mer oligonucleotides were synthessized in situ using Agilent inkjet SurePrint technology. Four arrays were printed on each 1 x 3-inch glass slide (…etc…) | |
| 8 | Technology Type | アレイをプリント/合成した手法。 | “in situ oligo features”, “spotted antibody features”, “spotted colony features”, “spotted ds DNA features”, “spotted protein features”, “spotted_ss_PCR_amplicon_features”, “spotted_ss_oligo_features” | in situ oligo features |
| 9 | Surface Type | 基質表面のコーティング方法(下から選択)。プローブはコーティングによって固定されます。 | polylysine, aminosilane, “other surface type” | polylysine |
| 10 | Substrate Type | マイクロアレイの素材/基質 | glass, nitrocellulose, nylon, silicon, “other substrate type” | glass |
| 11 | Sequence Polymer Type | プローブを構成するポリマー | DNA, RNA, protein | DNA |
| 12 | Term Source Name | クロスリファレンスするリソース名。例 Reporter Database Entry [uniprot]。 クロスリファレンスしたリソース名を [] に記載することで URL を辿ってリソースを特定することができます (下のヘッダー13を参照)。 | クロス参照リソースリスト | uniprot |
| 13 | Term Source File | Term Source Name (上のヘッダー12参照) に記載されたリソースがホストされている URL。 | 有効な URL | http://www.uniprot.org |
ADF テーブルセクション
ADF テーブルセクション中のヘッダーも controlled vocabulary を使います。GEA が提供している ADF テンプレートファイルには正しいヘッダーが含まれているのでゼロから作成する必要はありません。テンプレート中のヘッダーは編集しないでください。
- composite_element_array_ADF_template.xls
- commercial_array_header_template.xls
- reporter_only_array_ADF_template.xls
- spotted_array_ADF_template.xls
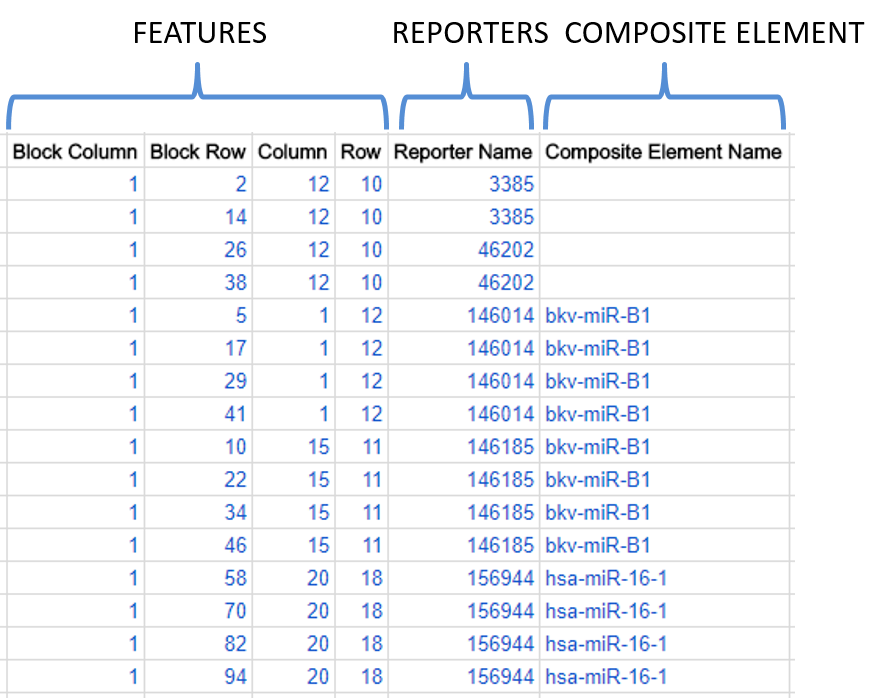
Feature、reporter と composite element
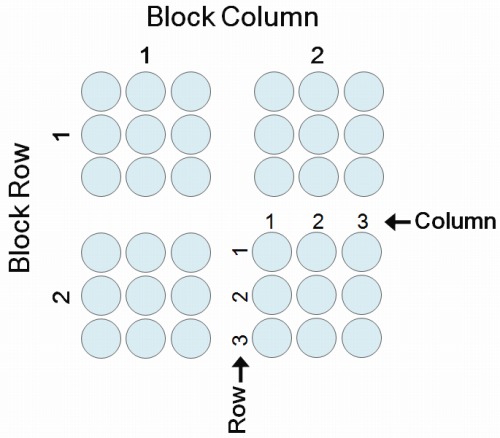
Feature (スポットアレイのみ):
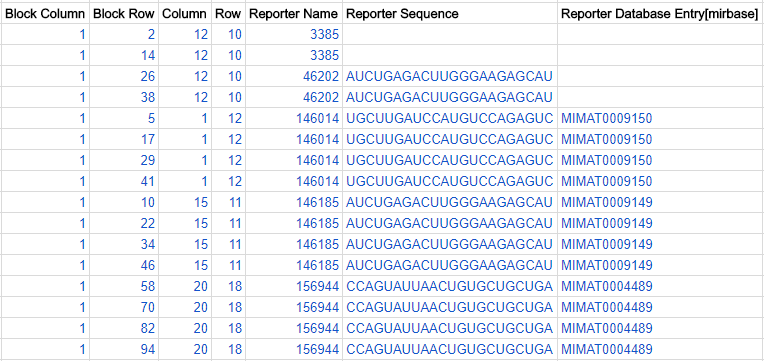
アレイ上の各スポットを feature と呼びます。Feature の位置は四つの座標 Block Column、Block Row、Column と Row で示します。これらの四座標はマイクロアレイ上のユニークな位置に対応しているため feature が重複することはありません。 Control spot のような何もスポットされていないスポットも含め全ての feature を ADF ファイルで記載してください。 以下に四座標の図式を示します。
Reporter (全てのアレイで必須):
Reporter は通常 “probe” と呼ばれているものです。各 reporter は特定の塩基配列と role (experimental か control) を持ちます。 原則として reporter にはユニークな名前を付けます。同一配列のプローブがアレイの異なるスポット (feature) にプリントされている場合は異なる座標で同じ reporter name が繰り返されることになります。
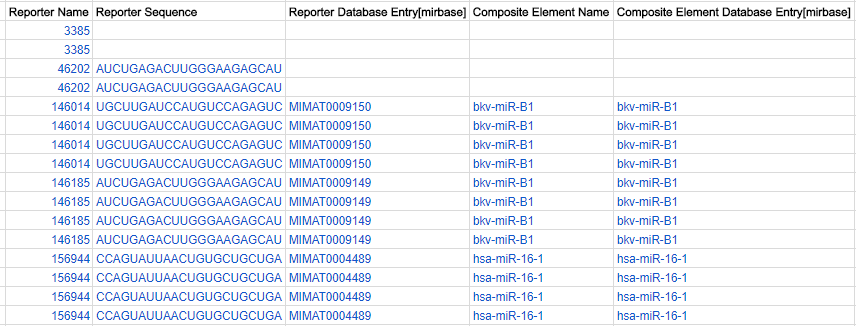
Composite element (全てのアレイにおいて任意):
Composite element で reporter 同士の関係性、同じ遺伝子や転写産物由来であるといったこと、などの情報を付加することができます。 例えばある遺伝子の二つのエキソンから異なる reporter がデザインされている場合、これらの reporter が同じ composite element(遺伝子)に属していることを示すことができます(composite element の name がユニークである場合)。 Affymetrix アレイは probe set で対象を測定する仕組みであるため、その ADF ファイルは常に composite element を持ちます。
以下に16個の feature、5個の reporter と2個の composite element を持つ仮想的な例を示します。
Reporter のアノテーション
Reporter の塩基配列、外部データベースエントリのアクセッション番号 (例 reporter の基になった cDNA 配列の RefSeq アクセッション番号) やリファレンスゲノム上の位置を付加して reporter をアノテーションします。塩基配列には Reporter Sequence カラムを使い、外部エンティティやゲノム座標参照は1つ以上の Reporter Database Entry [xxx] カラムを使います。
マイクロアレイの異なる場所に同じ reporter がプリントされているケースなど ADF テーブルで reporter が複数回登場する場合、Rerpoter に付与されているアノテーション情報は ADF テーブルで一致している必要があります。
ブランクコントロールなどクロスリファレンスが無い reporter のアノテーションは空欄にします。キュレータがアノテーション情報のフォーマットと値をチェックするため ”NA”、”empty” や “unmapped” 等のカスタム値を記入しないでください。
Reporter 配列:
全てのアレイ(ペプチドアレイを含む)の配列は single-letter IUPAC code で記載します。 全ての experimental reporter と “array control biosequence” type の control reporter は配列アノテーションが付与されている必要があります。 一方、何もスポットされていないバッファーのみ、ラベルのみの control reporter にアノテーションを付与してはいけません。experimental と control reporter については下のセクションをご覧ください。
外部データベースエントリの参照:
参照したい外部データベースが許容されているリソースリストにあるかどうかを探し、ある場合は Database Entry [xxx] の [] にリソース名を記入します。そしてカラム値として参照エントリのアクセッション番号を記入します。複数アクセッション番号はセミコロン ; で区切って記載します(例 AJ12345;BX45678)。 異なる参照先データベースによるアノテーションの場合は別々の Reporter Database Entry [xxx] カラムを使います。ADF ヘッダーの Term Source Name と Term Source File フィールドにデータベースの名前と URL を記載します。
引用したいデータベースがリストに無い場合はデータベースの URL を GEA チームに連絡します。
アクセッション番号の方がフリーテキストの名前よりも安定で追跡可能であるため、エントリへのリンクは名前ではなくアクセッション番号に依存しています。加えて各データベースはアクセッション番号のフォーマットを定めているためコンピュータによる解析が容易です。例えば NCBI RefSeq curated cDNAs は NM_ に数字が続くフォーマットのアクセッション番号形式になっています。
ゲノムマップ上の位置:
データベースの略称の代わりにゲノム座標上の位置を chromosome_coordinate に繋げて Reporter Database Entry [xxx] の [] に記入することができます(例 Reporter Database Entry [chromosome_coordinate:GRCh38])。正式な公共データベース(ENA、GenBank や DDBJ)で使われているリファレンスゲノム名、例えば GRCh38、を使うようにします。ゲノム上の座標は chrName:start position-end position 形式で記載します(例 chr1:1234-5678)。
下記は仮想的な reporter アノテーションの例です。同一データベースの複数アクセッション番号をセミコロンで区切って列挙しています。
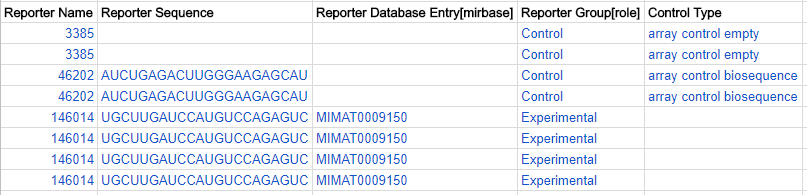
Repoter role - experimental もしくは control
各 Reporter の role、experimental もしくは control、を Reporter Group [role] カラムに記入します。
各 control reporter の種類を Control Type カラムに記入します(experimental reporter はこのカラムに値を記入してはいけません)。Experimental Factor Ontology (EFO) における control type に記載できる語彙は以下です。
| 語句 | 意味 |
|---|---|
| array control biosequence | ヒトのマイクロアレイにおける E. coli のスパイク配列。Reporter Sequence が必須。 |
| array control buffer | アレイにスポットされたバッファー。Reporter Sequence は空にします。 |
| array control empty | 何もアレイにスポットされていない(ブランク)。Reporter Sequence は空にします。 |
| array control genomic DNA | 例 サケ精子 DNA |
| array control label | Landing light。Reporter Sequence は空にします。 |
| array control reporter size | サイズスタンダード。 |
| array control spike calibration | 例 いくつかの濃度で導入されたスパイク配列 |
| array control design | 上記で適用な語彙が無い場合(例 control でそれ以上の情報が存在しない場合)。 |
ADF テーブルでの reporter role の例。
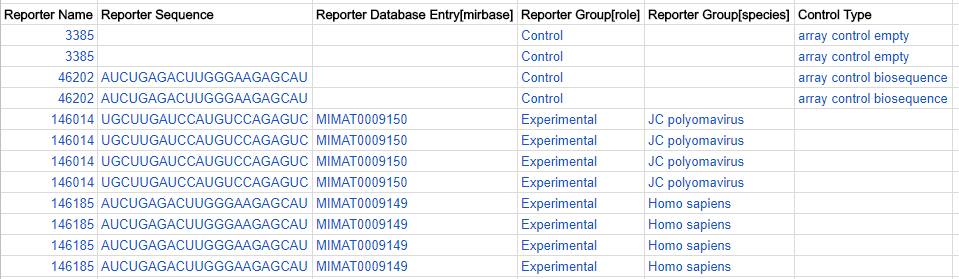
生物種による reporter grouping(複数生物種 ADF の場合のみ)
アレイが一つ以上の生物種からデザインされている場合、由来する生物種を Reporter Group [species] カラムで示すことができます。NCBI Taxonomy に登録されている学名を記載します。例 Homo sapiens。
ウイルスとヒトの microRNA 配列から構成されるアレイの例。
Composite element のアノテーション
Composite element のアノテーションは Composite Element Database Entry [xxx] カラムを使って外部データベース参照によって記述します。加えて Composite Element Comment カラムを Composite element をコメント注釈することに使うことができます。