DDBJ Annotated/Assembled Sequences
メタゲノムアセンブリ
微生物は生物多様性の多くの部分を占めていますが、生息環境が多様であることからそれらの多くは培養することができず、従来の培養を経たゲノム解析で得られる知見は限られています。メタゲノム解析とは培養に依存することなく網羅的に微生物のゲノムを解析する手法であり、培養することができない微生物種について多くの知見をもたらしています。
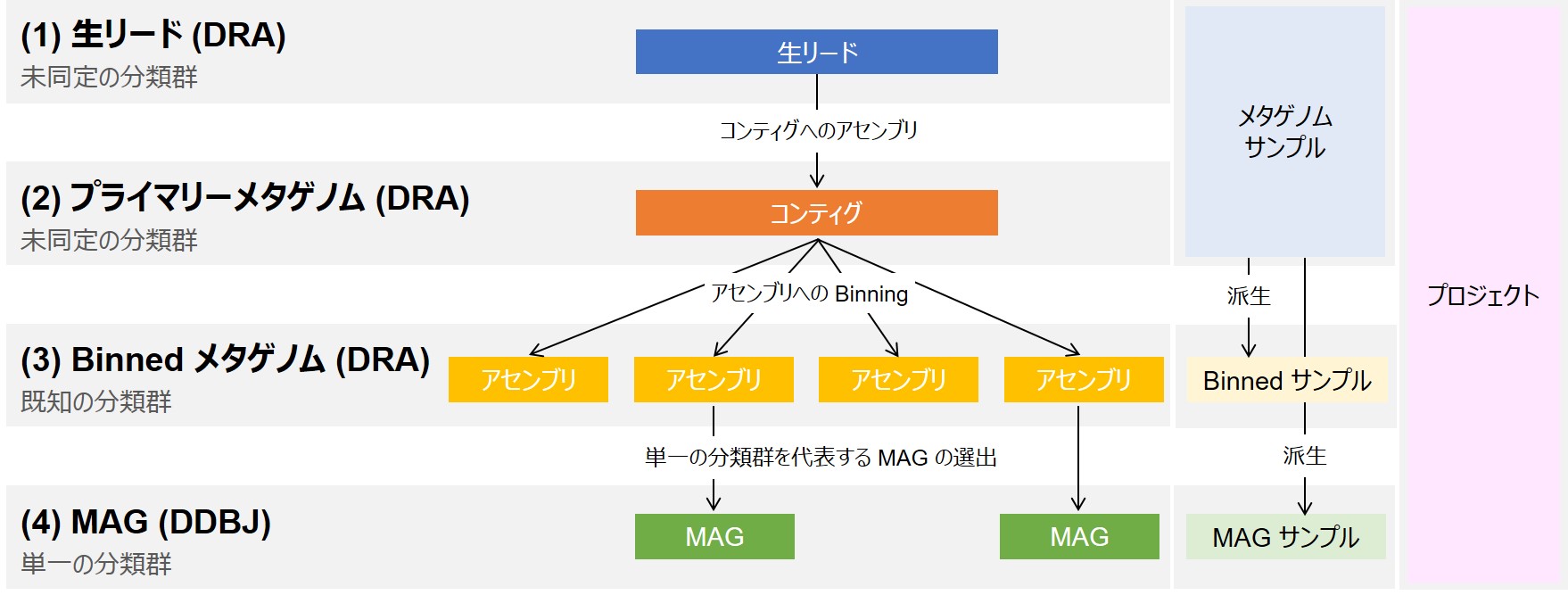
メタゲノムプロジェクトは解析段階に応じたいくつかのデータから構成されており、アセンブリ段階に応じて四つに区分されます。 (1) アセンブル前の NGS 生リード (2) 分類群が未同定のアセンブルされたコンティグ配列(プライマリーメタゲノム) (3) Binning され既知の分類群に帰属されたアセンブリ配列(Binned メタゲノム) (4) 単一の分類群に帰属された Binned アセンブリ配列の中で、分類群を代表する、配列の完成度・コンタミネーション割合といった指標が最も高品質なアセンブリ配列(Metagenome-Assembled Genome、MAG)
DDBJ センターにおいて (1)-(3) は DRA で、(4) は DDBJ で受付けています。MAG 配列の品質と区分に関してはこちらの文献を参照してください。 配列が由来するサンプル情報に関して、(1)-(2) はメタゲノムサンプルを、(3) と (4) はメタゲノムサンプルから派生した Binned と MAG サンプルをそれぞれ BioSample に登録します。 研究概要は BioProject に登録し、全てのデータから同じ BioProject を参照することで、一連のデータをメタゲノムプロジェクトとしてまとめます。
本ページでは各段階のデータを BioProject/BioSample/DRA/DDBJ へ登録する方法を説明しています。 DRA への生データの登録は原則として必須です。
メタゲノムアセンブリデータの登録
(1) 生リード
メタゲノムプロジェクトの生リードは DRA Run に登録します。
BioProject
メタゲノム/環境サンプルプロジェクトとして登録します。 生物名は metagenome organism names から適切なものを選び、”xyz metagenome” (例 soil metagenome) を記載します。
BioSample
MIxS MIMS.me パッケージを選択します。 メタゲノムサンプルの場合、生物名は metagenome organism names から適切なものを選び、”xyz metagenome” (例 soil metagenome) を記載します。サンプルに関する詳細情報を記載します。
DRA
生シークエンスデータを DRA Run に登録します。
(2) プライマリーメタゲノム
生リードをアセンブルしたコンティグ配列を DRA Analysis に登録します。
BioProject
(1) 生リードと同じです。
BioSample
(1) 生リードと同じです。
DRA
DRA に登録する場合、プライマリーメタゲノム配列を fasta/bam ファイルで (1) の生リードが登録された DRA Run とセットで Analysis に登録(Analysis type = “De Novo Assembly”)します。DRA 登録用エクセル を使い、解析ソフトウェアの情報を Analysis step に記載します。アセンブリの品質に関する completeness や contamination といった指標も Attributes に含めることができます。
DRA ウェブ登録システムを使って Analysis を登録する場合、Analysis description に以下の形式で参照している BioSample アクセッション番号 (必須)、解析ステップ (必須)、及び、アセンブリの品質に関する指標 (任意) を記載します。
- BioSample: SAMD00000001
- Analysis step: canu 2.1, pilon 1.24, CheckM 1.1.3
- Quality: completeness 85.3, contamination 0
Analysis は ENA/NCBI と共有されません。また、DDBJ Search でインデックスされず、ftp でメタデータ XML とデータファイルが公開されるのみとなります(例 DRZ000001)。
(3) Binned メタゲノム
Binning され、既知の分類群に帰属されたアセンブリ配列を DRA Analysis に登録します。
BioProject
(1) 生リードと同じです。
BioSample
Binned サンプル用に仮想的なサンプルを登録します。パッケージは “MIMAG” を選択し、生物名には uncultured が冠されていない、Binned アセンブリが由来する生物名を記載します (例 “Agrobacterium tumefaciens”、Agrobacterium sp.”、”Rhizobiaceae bacterium”)。 (1) の DRA Run 登録に使用する “xyz metagenome” メタゲノムサンプルとは別に仮想的な Binned 用サンプルが必要になります。
解析ツール GTDB が割り当てた生物名で NCBI Taxonomy に登録されていない名前は、そのまま使うことができません。対応する NCBI Taxonomy に登録されている名前に変換した上で登録します。
由来サンプルを示すため以下の属性を記載します。
メタゲノムの由来を metagenome organism names から適切なものを選び metagenome_source に “xyz metagenome” (例 soil metagenome) として記載します。 例 metagenome_source: soil metagenome
仮想的な BioSample の派生元である (1) で登録したメタゲノムサンプルのアクセッション番号を derived_from に記載します。 例 derived_from: SAMD00000001 derived_from: SAMD00000002,SAMD00000003,SAMD00000010-SAMD00000015
DRA
DRA に登録する場合、Binned アセンブリ配列を fasta/bam ファイルで、(1) の生リードが登録された DRA Run とセットで Analysis に登録します。DRA 登録用エクセル を使い、Analysis type = “De Novo Assembly” を選択し、解析ソフトウェアの情報を Analysis step、アセンブリの品質や binning に関する指標を Attributes に記載します。
DRA ウェブ登録システムを使って Analysis を登録する場合、Analysis description に参照している仮想的な BioSample アクセッション番号 (必須)、解析ステップ (必須)、及び、アセンブリの品質に関する指標 (任意) を以下のように記載します。
- BioSample: SAMD00000001
- Analysis step: canu 2.1, pilon 1.24, CheckM 1.1.3
- Quality: completeness 85.3, contamination 0
Analysis は ENA/NCBI と共有されません。また、DDBJ Search でインデックスされず、ftp でメタデータ XML とデータファイルが公開されるのみとなります(例 DRZ000001)。
(4) MAG
単一の分類群に帰属された Binned アセンブリ配列の中で、分類群を代表する、配列の完成度・コンタミネーション割合といった指標が最も高品質なアセンブリ配列(Metagenome-Assembled Genome、MAG)は DDBJ の ENV division にゲノムエントリとして登録します。MAG 配列の品質と区分に関してはこちらの文献を参照してください。
BioProject
(1) 生リードと同じです。
BioSample
MAG サンプル用に仮想的なサンプルを登録します。パッケージは “MIMAG” を選択し、生物名には uncultured が冠されていない、MAG 配列が由来する生物名を記載します (例 “Agrobacterium tumefaciens”)。 (1) の DRA Run 登録に使用する “xyz metagenome” メタゲノムサンプルとは別に仮想的な MAG 用サンプルが必要になります。
解析ツール GTDB が割り当てた生物名で NCBI Taxonomy に登録されていない名前は、そのまま使うことができません。対応する NCBI Taxonomy に登録されている名前に変換した上で登録します。
由来サンプルを示すため以下の属性を記載します。
メタゲノムの由来を metagenome organism names から適切なものを選び metagenome_source に “xyz metagenome” (例 soil metagenome) として記載します。 例 metagenome_source: soil metagenome
仮想的な BioSample の派生元である (1) で登録したメタゲノムサンプルのアクセッション番号を derived_from に記載します。 例 derived_from: SAMD00000001 derived_from: SAMD00000002,SAMD00000003,SAMD00000010-SAMD00000015
DDBJ
MAG は ENV division のゲノムエントリとして登録します。 MSS が登録受付窓口になります。 MAG 登録では source feature に以下の Qualifier が必須になります。
MAG 特有のルールとして必須
- /metagenome_source = “xyz metagenome”(”xyz metagenome” は metagenome organism names に含まれていること)を記載。
ENV Division エントリとして必須
全てのエントリで必須
また、ゲノムエントリとして ST_COMMENT に以下のアセンブリ情報が必須になります。
- Assembly Method
- Genome Coverage
- Sequencing Technology
- Assembly Name (真核生物の場合に必須)
MAG エントリ(ENV division)では /strain を記載することはできません。
また、宿主の情報がある場合は source feature に /host で記載します。