DDBJ Annotated/Assembled Sequences
UME ユーザーマニュアル
UME (Utilities for MSS file Error check) は Mass Submission System (MSS) を利用して DDBJ に登録する際に必要な 配列ファイル とアノテーションファイル のフォーマットチェックと、それらファイルの記載を元に CDS feature のアミノ酸翻訳チェックを行うGUIツールです。Parser と transChecker を内包しています。
基本機能
| 名称 | 機能 |
|---|---|
| Parser | 配列ファイル とアノテーションファイル の構文とフォーマットを検証します。 登録ファイルを DDBJ 送付する際には、必ず Parser を用いてファイルの確認を行い、エラーのない状態にしてから送付してください。 |
| transChecker | CDS (タンパク質をコードする配列)feature でアミノ酸翻訳の可否とその配列を検証します。 |
| FFconv | 配列ファイル と アノテーションファイル から、擬似的な フラットファイル を作成します。 |
| AGPParser | contig(CON エントリ) を登録する場合、AGPファイル と アノテーションファイル の構文とフォーマットを検証します。 |
インストール
UME パッケージを MSS データファイルチェック用ツール からダウンロードし、以下の点に注意して、解凍してください。
その後、UME フォルダ内にある UME アイコンをダブルクリックして起動します。
- Windows: zip ファイルの展開表示
- Windows では、zip ファイルが圧縮状態のままでも、フォルダのように閲覧可能な場合があります。 UME 実行時には、ファイルを解凍しておく必要がありますので、ご注意ください。
- unix/macOS: ファイルの解凍方法
- 解凍ソフトを使用するか、以下のコマンドを用いて解凍してください。
$ unzip UME_unix_<Version No.>.zip
- macOS: ファイル名とフォルダ名について
- 配列ファイル、アノテーションファイル、AGP ファイル のファイル名またはフォルダ名に、マルチバイト文字が含まれていますと一部のバージョンの macOS では正常に動作しない場合がありますので、ファイル名とフォルダ名にマルチバイト文字を混在させないようにご注意ください。
初期設定
UME の起動
UME フォルダ内にある UME アイコン(ご使用の PC の環境設定に依存して何らかの拡張子が表示されることがあります)をダブルクリックして起動します。
macOS の場合、UME アイコンをクリックしても起動できないことがあります。
その場合はターミナルから下記のようにコマンド実行してください。
$ cd UME # UME を展開したディレクトリに移動してください。
$ ./UME.sh
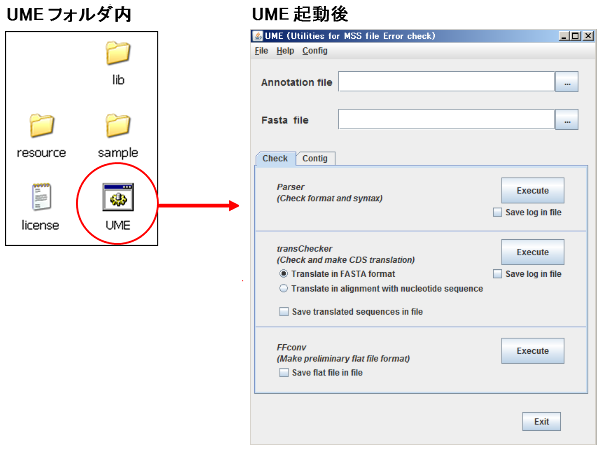
- lib フォルダ
- UME が使用するライブラリファイルがあります。
正常に動作しなくなる恐れがありますので、絶対に内部は変更しないでください。
移動もしないでください。 - resource フォルダ
- UME が使用するリソースファイルがあります。
正常に動作しなくなる恐れがありますので、絶対に内部は変更しないでください。
移動もしないでください。 - sample フォルダ
- DDBJ 登録用のサンプルファイルがあります。
- UME ファイル
- ダブルクリックすると UME が起動します。
- license ファイル
- 使用許諾に関するテキストファイルです。ご一読ください。 内容は 使用許諾と同じです。
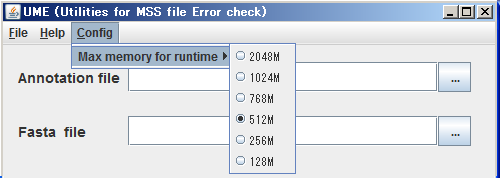
メモリの初期設定
UME を起動しましたら、メモリ割り当てサイズを調節します。
デフォルトは 512 Mbytes ですが、ご使用の PCのスペックに合わせて変更してください。
(1) 画面上部のメニューから Config をクリックします。
(2) Max memory for runtime のプルダウンメニューから、クリックして選択します。
登録用ファイルの検証
Check パネルの構成
下図で赤い四角で囲んだ部分にある Check タブをクリックしますと、この表示になります。
UME を起動後、最初に開いているパネルです。
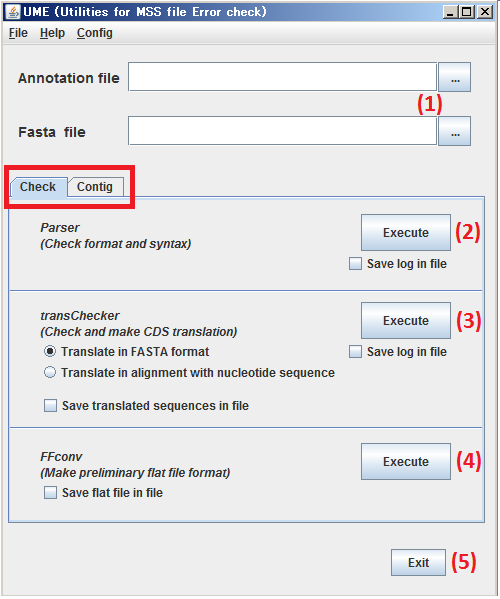
(1) 処理対象とする アノテーションファイル と配列ファイル (Fasta File)をそれぞれ指定します。
(2) Execute をクリックしますと、(1) で指定したファイルを Parser で検証します。
(3) Execute をクリックしますと、(1) で指定したファイルから transChecker でアミノ酸翻訳を確認します。
(4) Execute をクリックしますと、(1) で指定したファイルから FFconv で擬似フラットファイルを作成します。
(5) Exit をクリックしますと、UME を終了します。
Parser: 構文チェックツール
以下の手順で Parser による登録ファイルの構文チェックを実行してください。
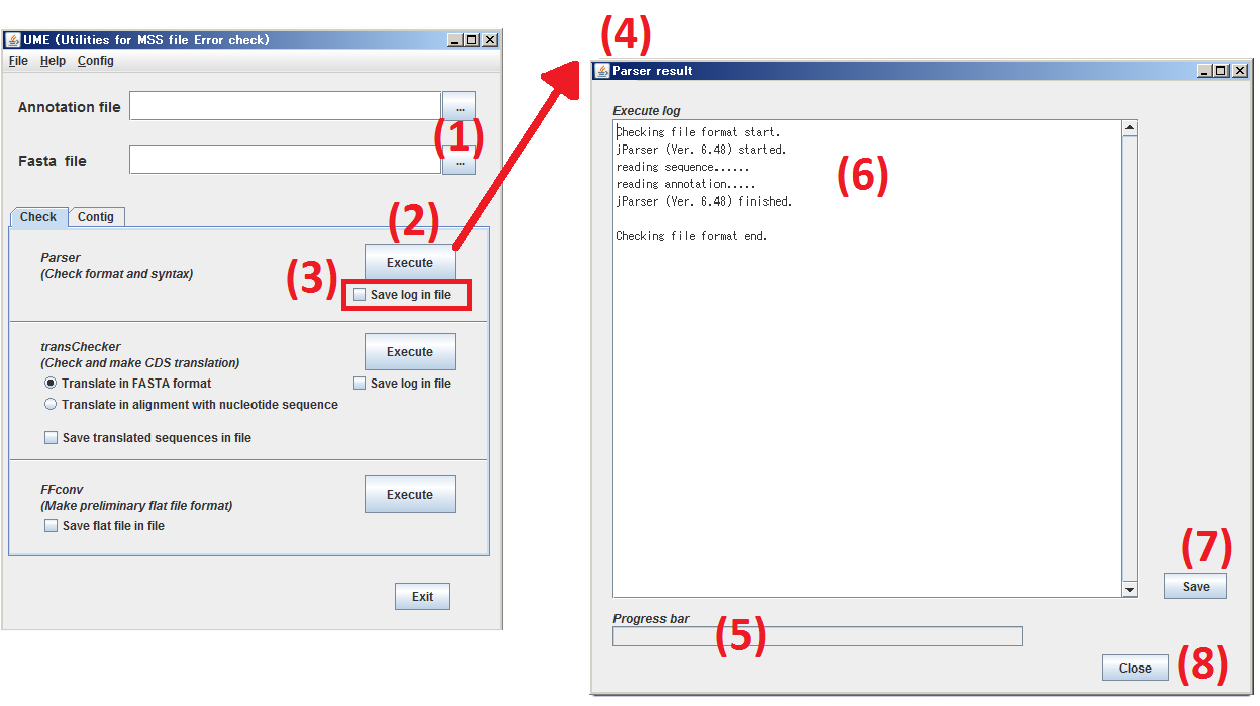
Check タブをクリックし、Check パネルを表示させます。
(1) 処理対象とする アノテーションファイル と配列ファイル をそれぞれ指定します。
(2) Execute をクリックしますと、(1) で指定したファイルを Parser でチェックします。
(3) Save log in file をチェックした場合は、Parser の実行結果をテキストファイルに保存します。
(4) Parser の実行結果表示ウィンドウが開きます。
(5) Parser チェックの進行状況を表示します。
(6) Parser チェックの実行結果を表示します。
(7) Save をクリックしますと、(6) の内容をテキストファイルに保存します。
(8) Close をクリックしますと、この結果表示ウィンドウを閉じます。
Parser は、エラーが発生している場合、エラーメッセージを出力します。
エラーメッセージの意味は Parser エラーメッセージ をご覧ください。
transChecker: アミノ酸翻訳検証ツール
登録データ中に CDS feature を含む場合 transChecker によるアミノ酸翻訳チェックをお願いします。
ただし、transChecker を実行する前に、Parser でエラーが出力されない状態にしておく必要があります。
Parser でエラーが検出される場合は、transChecker を実行することが出来ませんのでご注意ください。
以下の手順で transChecker によるアミノ酸翻訳チェックを実行します。
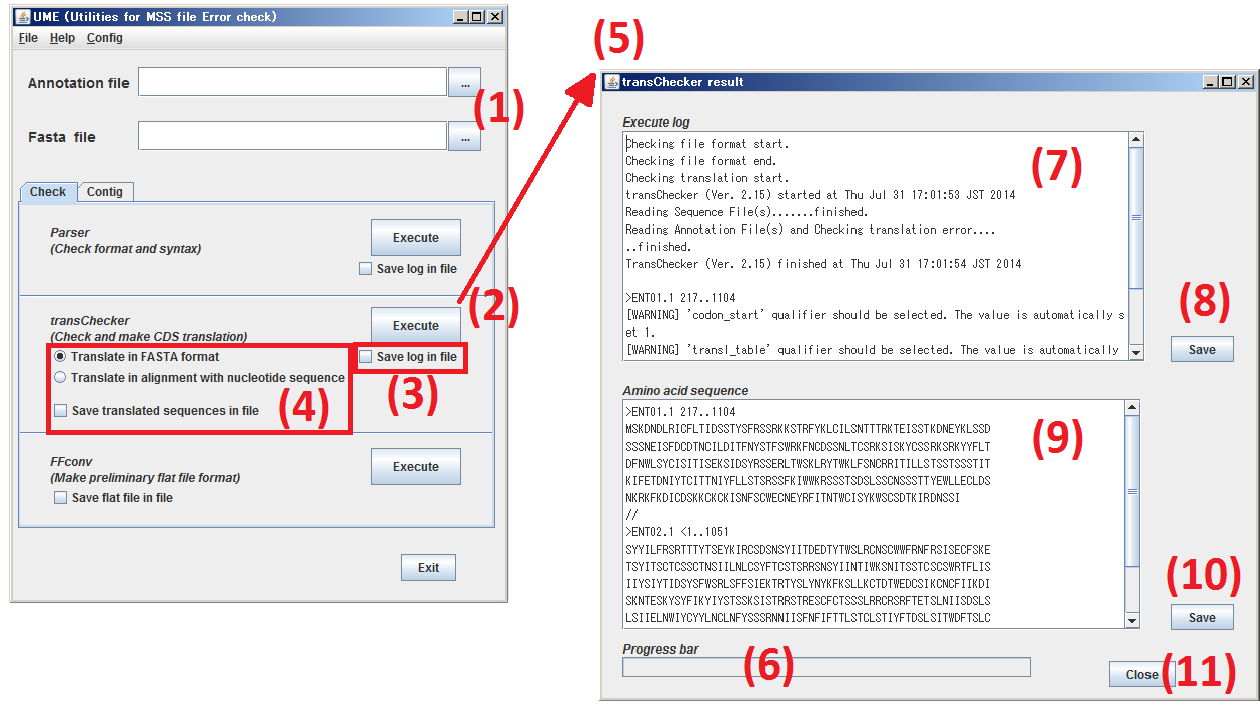
Check タブをクリックし、 Check パネル を表示させます。
(1) 処理対象とする アノテーションファイル と 配列ファイル をそれぞれ指定します。
(2) Execute をクリックしますと、(1) で指定したファイルを transChecker でチェックします。
(3) Save log in file をチェックした場合は、transChecker の実行結果をテキストファイルに保存します。
(4) Save translated sequences in file をチェックした場合、transChecker 実行時に出力される
翻訳アミノ酸配列を選択された書式でテキストファイルに保存します。出力書式は以下の2通りです。
| FASTA-like format | Translate in FASTA format を選択した場合 |
| 塩基配列とのアラインメント | Translate in alignment with nucleotide sequence |
(5) transChecker の実行結果表示ウィンドウが開きます。
(6) transChecker チェックの進行状況を表示します。
(7) transChecker チェックの実行結果、エラーメッセージなどを表示します。
(8) Save をクリックしますと、(7) の内容をテキストファイルに保存します。
(9) transChecker チェックの実行結果、翻訳アミノ酸配列を (4) で選択した書式で表示します。
(10) Save をクリックしますと、(9) の内容をテキストファイルに保存します。
(11) Close をクリックしますと、この結果表示ウィンドウを閉じます。
transChecker は、エラーが発生している場合、エラーメッセージを出力します。
エラーメッセージの意味は transChecker エラーメッセージ をご覧ください。
FFconv: 擬似フラットファイル作成ツール
FFconv を使用して アノテーションファイル と 配列ファイル を DDBJ フラットファイルに変換し確認することが可能です。
ただし、FFconv を実行する前に、Parser でエラーが出力されない状態にしておく必要があります。
Parser でエラーが検出される場合、FFconv を実行することが出来ませんので、ご注意ください。
FFconv は以下の手順で実行します。
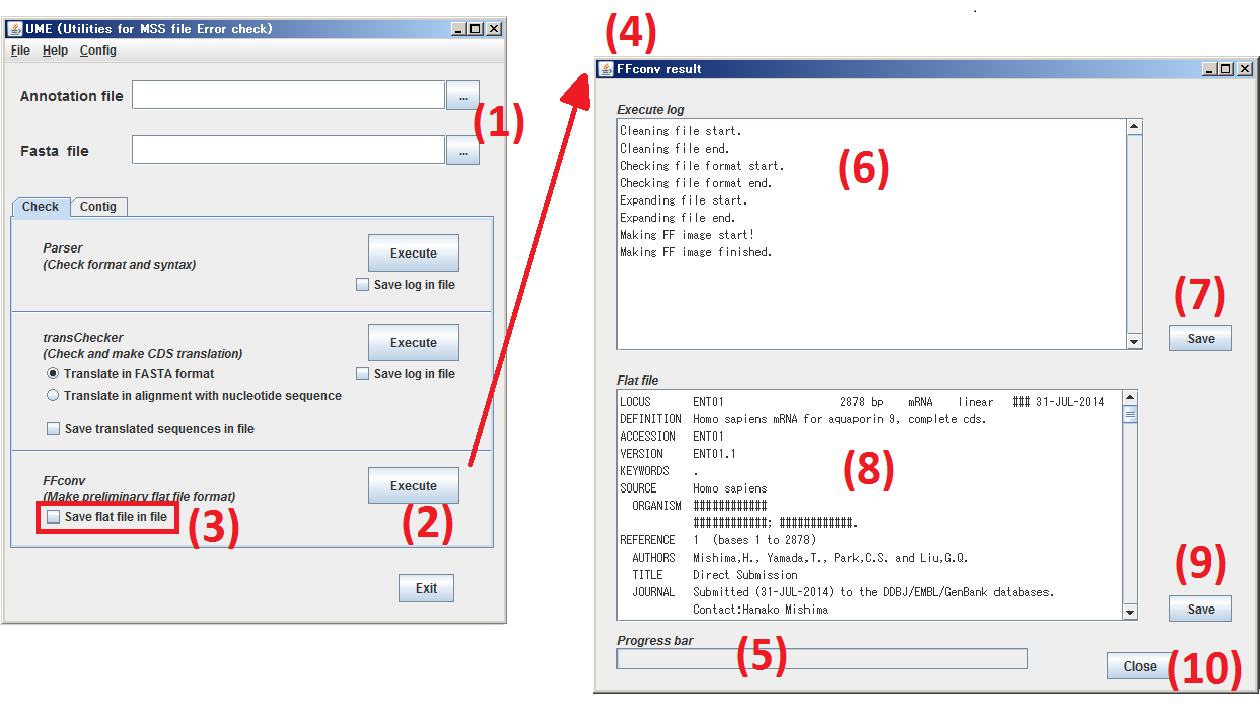
Check タブをクリックし、 Check パネル を表示させます。
(1) 処理対象とする アノテーションファイル と 配列ファイル をそれぞれ指定します。
(2) Execute をクリックしますと、(1) で指定したファイルから FFconv で擬似 DDBJ フラットファイルを作成します。
(3) Save flat file in file をチェックした場合は、FFconv の実行結果、擬似 DDBJ フラットファイルをテキストファイルに保存します。
(4) FFconv の実行結果表示ウィンドウが開きます。
(5) FFconv の進行状況を表示します。
(6) FFconv の実行結果を表示します。
(7) Save をクリックしますと、(6) の内容をテキストファイルに保存します。
(8) FFconv の実行結果、擬似 DDBJ フラットファイルを表示します。
(9) Save をクリックしますと、(8) の内容をテキストファイルに保存します。
(10) Close をクリックしますと、この結果表示ウィンドウを閉じます。
Contig 登録用ファイルの検証
[重要] CON データの新規登録は受け付けを終了しました。
Contig パネルの構成
下図で赤い四角で囲んだ部分にある Contig タブをクリックしますと、この表示になります。
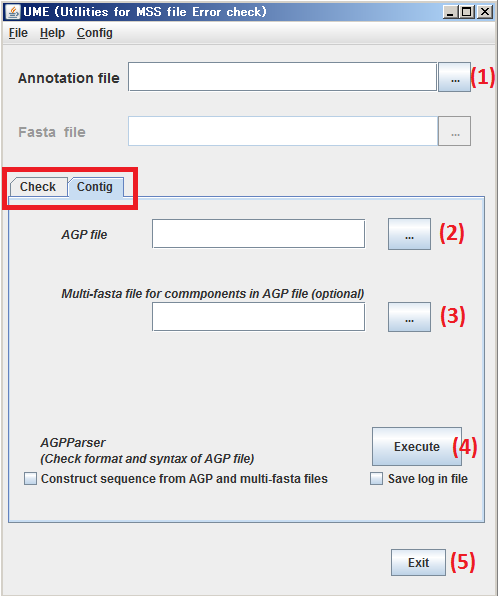
(1) 処理対象とする アノテーションファイル を指定します。
(2) 処理対象とする AGPファイル を指定します。
(3) (2) で引用の対象とする 塩基配列ファイル を指定します。
(4) Execute をクリックしますと、指定したファイルを AGPParser でチェックします。
(5) Exit をクリックしますと、UME を終了します。
AGPParser 実行
AGPParser は AGPファイル のフォーマットをチェックします。
アノテーションファイル または AGPファイルで引用する配列で構成される multi-fasta ファイルを指定した場合、相互の整合性をチェックします。
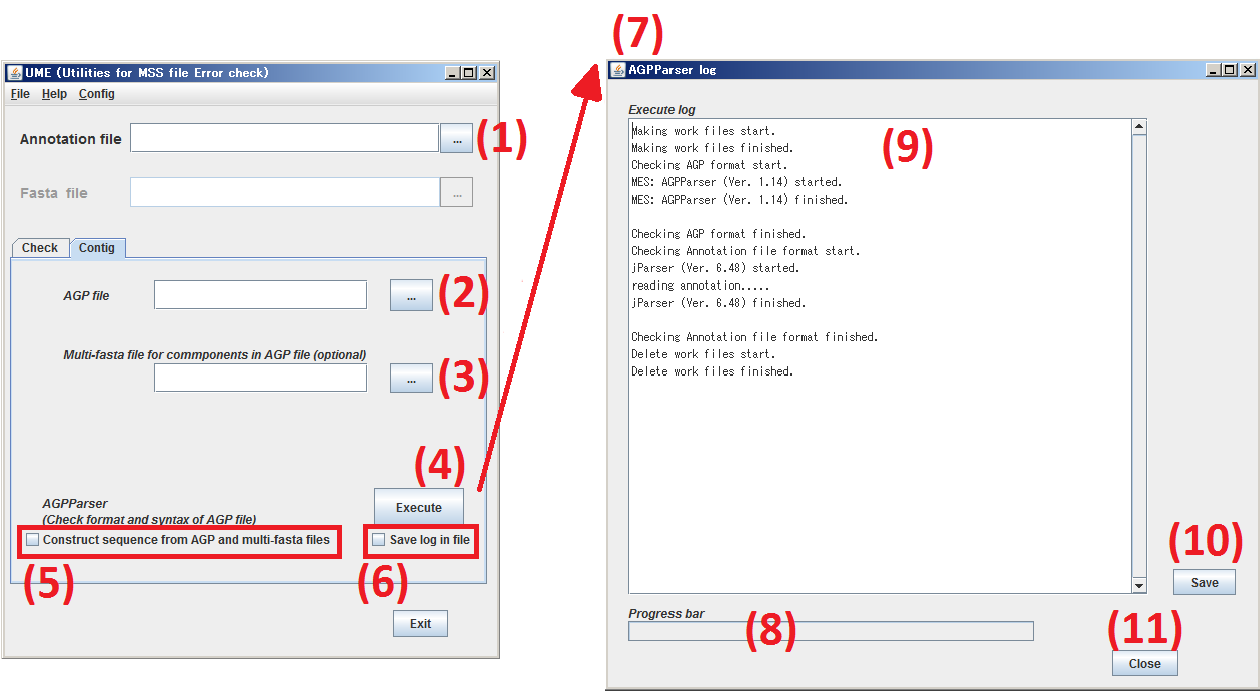
Contig タブをクリックし、 Contig パネル を表示させます。
(1) 処理対象とする アノテーションファイル を指定します。必須ではありません。
(2) 処理対象とする AGPファイル を指定します。必須です。
(3) 処理対象とする引用配列の 配列ファイル を指定します。必須ではありません。
(4) Execute をクリックしますと、(1)~(3) で指定したファイルを AGPParser でチェックします。
(5) [Construct sequence from AGP and multi-fasta files] をチェックした場合、2つのファイルの記載にしたがって
塩基配列を作成し、テキストファイルに保存します。
(6) Save log in file をチェックした場合は、AGPParser の実行結果をテキストファイルに保存します。
(7) AGPParser の実行結果表示ウィンドウが開きます。
(8) AGPParser の進行状況を表示します。
(9) AGPParser の実行結果を表示します。
(10) Save をクリックしますと、(9) の内容をテキストファイルに保存します。
(11) Close をクリックしますと、この結果表示ウィンドウを閉じます。
AGPparser はエラーが発生している場合、エラーメッセージを出力します。
エラーメッセージの意味は AGPparser エラーメッセージ をご覧ください。